Essa é uma revisão anterior do documento!
Tabela de conteúdos
Walmes Marques Zeviani
"Experiência não é o que acontece com você, mas o que você fez com o que lhe aconteceu." (Aldous Huxley)
"Feliz é aquele que transfere o que sabe e aprende o que ensina." (Cora Coralina)
"O que você sabe não tem valor, o valor está no que você faz com o que sabe." (Bruce Lee)
"Compreender que há outros pontos de vista é o início da sabedoria." (Campbell)
"Quem não sabe o que busca não identifica o que acha." (Immanuel Kant)
"Não sou aquele que sabe, mas aquele que busca." (Herman Hesse)
Engenheiro Agronômo pela Universidade Federal da Grande Dourados (2007), Mestre (2008-2009) e Doutor (2010-2013) em Estatística & Experimentação Agropecuária pela Universidade Federal de Lavras. Doutorando do Programa de Pós-Graduação em Estatística & Experimentação Agropecuária - Professor Assistente I do Departamento de Estatística da Universidade Federal do Paraná (aprovado por Concurso e contratado em Julho 2010) onde atualmente leciona.

========================================================================== Walmes Marques Zeviani LEG (Laboratório de Estatística e Geoinformação, 25.450418 S, 49.231759 W) Departamento de Estatística - Universidade Federal do Paraná Edifício da Administração - 3º andar - Centro Politécnico Curitiba - PR CEP: 81.531-990 Bairro: Jardim das Américas Caixa Postal: 19.081 fone: (+55) 41 3361 3573 VoIP: (3361 3600) 1053 1173 e-mail: walmes@ufpr.br twitter: @walmeszeviani homepage: http://www.leg.ufpr.br/~walmes linux user number: 531218 ==========================================================================
Dissertação de Mestrado
Avaliação de modelos de regressão não linear na cinética de liberação de potássio de resíduos orgânicos

Resumo: O potássio (K) é um elemento químico muito exigido pelas culturas, porém, sua ocorrência e disponibilidade em alguns solos brasileiros são inferiores à demanda pelas plantas. Seu fornecimento também pode ser feito a partir de fontes orgânicas de maneira eficiente, quando se conhece o padrão de liberação do nutriente. Modelos não lineares são adequados nessas situações, uma vez que estimam quantidades de interesse prático e apresentam boa qualidade de ajuste. Embora seu processo inferencial seja baseado em argumentos assintóticos, existem meios de se conhecer a intensidade da não linearidade. Neste trabalho avaliou-se a não linearidade, por meio das curvaturas de Bates & Watts, vício de Box e estudo das propriedades amostrais dos estimadores de mínimos quadrados por simulação, de dois modelos de regressão não linear. Tais modelos estimam o conteúdo de K prontamente liberável, o tempo de meia vida para a sua liberação e a taxa de liberação do K de lenta liberação. Os dados são provenientes do estudo, ao longo do tempo, da liberação de K de 11 resíduos orgânicos incubados em combinação com 3 tipos de solo. O modelo exponencial apresentou estimativas mais precisas para os parâmetros que o quociente e maior aproximação entre os resultados assintóticos e obtidos por simulação. O modelo exponencial foi mais adequado, em termos inferenciais e para aplicação prática, uma vez que, por todas as medidas, apresentou menor não linearidade.
Palavras-chaves: Curvatura de Bates e Watts. Vício de Box. Bootstrap. Tempo de meia vida.
Banca de Defesa:
Prof. Dr. Joel Augusto Muniz (Orientador) - UFLA
Prof. Dr. Carlos Alberto Silva (co-orientador) - UFLA
Profa. Dra. Taciana Villela Savian - UFLA
Prof. Dr. Luiz Alberto Beijo - UNIFAL-MG
Aprovada em 18 de setembro de 2009
Tese de Doutorado
Título: Parametrizações interpretáveis em modelos não lineares

Capítulo 1: PROCEDIMENTO PARA OBTER PARAMETRIZAÇÕES INTERPRETÁVEIS EM MODELOS NÃO LINEARES. Modelos de regressão não linear são considerados quando existe algum conhecimento preliminar sobre a relação entre variáveis. Tal conhecimento pode ser a respeito da própria natureza dos dados, uma equação diferencial e até mesmo a forma do diagrama de dispersão entre as variáveis. Em geral, seus parâmetros têm interpretação. Além disso, parâmetros de interesse, expressos como função dos parâmetros do modelo, são alvos de investigação. Para isso, o método delta, simulação Monte Carlo e procedimentos bootstrap são procedimentos adotados para fazer inferência. Além disso, uma reparametrização pode ser aplicada ao mo- delo de forma a representar esses parâmetros de interesse. Além de melhorar a interpretação do modelo, a presença do parâmetro alvo estende as possibilidades com relação a especificação de modelos e inferência estatística. O objetivo com esse trabalho é sistematizar o procedimento de aplicar reparametrizações. Ênfase é dada em modelos não lineares considerados em Ciências Agrárias. Uma lista com 17 modelos reparametrizados é fornecida. Breve discussão sobre os métodos de inferência é feita.
Capítulo 2: MODELO NÃO LINEAR PARA O NÍVEL DE DANO ECONÔMICO DA DESFOLHA NO ALGODOEIRO. O efeito da desfolha sobre a qualidade e produtividade das culturas é informação fundamental para definir estratégias de manejo, como intensidade e frequência de pastejo e colheita até o estabelecimento de níveis de dano econô- mico de forma a auxiliar decisões sobre o controle de pragas desfolhadoras. Para a cultura do algodão, assim como para outras tantas, a redução da produção pela desfolha pode ser representada por uma função não linear monótona não crescente. Diversos modelos podem satisfazer essa restrição, no entanto, existe a preocupa- ção de inferir sobre o nível de dano econômico, θq , pelo ajuste de um modelo. Dados de produção-desfolha do algodoeiro em função do estágio fenológico são considerados para inferir sobre o nível de dano econômico com os seguintes ob- jetivos: 1) propor uma parametrização de modelo que representasse o parâmetro, 2) avaliar parametrizações alternativas por meio de medidas de não linearidade, 3) aplicar inferência baseada em verossimilhança, 4) selecionar um modelo para descrever a relação entre produção e desfolha do algodoeiro em função do estágio fenológico. O modelo reparametrizado apresentou menores medidas de não linea- ridade nos estágios fenológicos com pronunciada relação não linear. Nos restantes, as medidas de curvatura, as correlações dos estimadores e os gráficos de perfil de verossimilhança indicaram que um sub-modelo deveria ser considerado.
Caítulo 3: PARAMETRIZAÇÃO DO MODELO VAN GENUCHTEN PARA INFERÊNCIA SOBRE OS PARÂMETROS S E I. A água é indispensável para produção das culturas pois está envolvida no transporte de nutrientes, reações químicas, processos físicos e manutenção da vida do solo. O conhecimento sobre a curva de rentenção de água (CRA) do solo é fundamental para estabelecer estratégias de manejo. A qualidade física do solo é depende da CRA e os parâmetros I, tensão do ponto de inflexão da CRA, e S , taxa de variação no ponto de inflexão, considerados como indicadores da quali- dade física, são parâmetros relacionados a medidas descritivas da distribuição do tamanho de poros do solo. Com este trabalho, objetiva-se verificar o efeito da po- sição de amostragem e profundidade do solo sobre os parâmetros I e S da CRA. Para isso 1) considerou-se ANOVA simples e 2) ANOVA ponderada pela variância das estimativas desses parâmetros em cada unidade experimental em comparação com 3) o uso de modelos não lineares de efeito misto em uma parametrização de- senvolvida para I e S . Nenhum dos métodos alternativos de análise foi superior ao modelo não linear de efeitos mistos na parametrização desenvolvida, que apresen- tou intervalos mais estreitos para estimativas dos parâmetros e apontou efeito de posição e profundidade de coleta nos parâmetros I e S.
Palavras-chaves: Verossimilhança. Método delta. Medida de curvatura. Efeitos mistos. van Genuchten.
Banca de Defesa:
Prof. Dr. Joel Augusto Muniz (Orientador) - UFLA
Prof. Dr. Paulo Justiniano Ribeiro Jr (co-orientador) - UFPR
Prof. Dr. Júlio da Motta Singer - IME-USP
Prof. Dr. Júlio Silvio de Sousa Bueno Filho - UFLA
Prof. Dr. Augusto Ramalho de Morais - UFLA
Aprovada em 13 de maio de 2013.
Ensino
Disciplinas já ofertadas e em andamento. Clique na turma para ir para a página da disciplina onde estão disponíveis o histórico das aulas, provas, gabaritos, listas de exercícios e notas.
| Disciplina | Curso | Turma | Ano | Semestre |
|---|---|---|---|---|
| Estatística Básica (CE-003 - Estatística II) | Administração | N2 | 2010 | 2º |
| Estatística Básica (CE-003 - Estatística II) | Administração | N3 | 2010 | 2º |
| Estatística Básica (CE-003 - Estatística II) | Engenharia Cívil | B | 2011 | 1º |
| Estatística Computacional (CE-223) | Estatística | Única | 2011 | 1º |
| Estatística Básica (CE-002 - Estatística I) | Agronomia | B | 2011 | 2º |
| Estatística Computacional I (CE-083) | Estatística | A e B | 2011 | 2º |
| Estatística Básica (CE-002 - Estatística I) | Agronomia | B | 2012 | 1º |
| Estatística Computacional I (CE-083) | Estatística | EST | 2012 | 1º |
| Controle de Processos Industriais (CE-074) | Estatística | EST | 2012 | 2º |
| Estatística Computacional I (CE-083) | Estatística | EST | 2012 | 2º |
| Estatística Computacional I (CE-083) | Estatística | EST | 2013 | 1º |
| Estatística Básica (CE-002 - Estatística I) | Agronomia | A | 2013 | 1º |
| Bioestatística (CE-001 - Bioestatística) | Biologia | MB | 2013 | 1º |
| Estatística Computacional I (CE-083) | Estatística | EST | 2013 | 2º |
| Estatística Computacional II (CE-089) | Estatística | EST | 2013 | 2º |
| Análise de regressão linear (CE-071) | Estatística | EST | 2014 | 1º (em andamento) |
| Estatística Computacional I (CE-083) | Estatística | EST | 2014 | 1º (em andamento) |
| Bioestatística (CE-001 - Bioestatística) | Biologia | MB | 2014 | 1º (em andamento) |
Cursos de R
| Tema | Público | Lugar | Período |
|---|---|---|---|
| Modelagem e análise de dados experimentais com o programa computacional R | Pós Graduação em Produção Vegetal - UFPR | UFPR, Curitba-PR | 7 à 11 de Fevereiro de 2011 |
| Curso de capacitação ao ambiente estatístico R | Docentes da Faculdade de Ciências Agrárias da Universidade Federal da Grande Dourados | UFGD, Dourados-MS | 25 à 29 de abril de 2011 |
| Estatística Experimental Aplicada | Pesquisadores da Embrapa Arroz e Feijão e acadêmicos da UFG | CNPAF, Santo Antônio de Goiás-GO | 07 à 11 de novembro de 2011 |
| Estatística Experimental Aplicada | Pesquisadores da Epagri-SC e Engenheiros Agronômos | IF de Videira, Videira-SC | 18 e 19 de maio de 2012 |
| Análise Estatística em Ambiente R | Curso de Especialização em Instrumentação Estatística da UNOCHAPECÓ | UNOCHAPECÓ, Chapecó - SC | 01-02/06 e 15-16/06 de 2012 |
| Curso em análise de experimentos com afastamento das pressuposições | Pesquisadores da Embrapa Agropecuária Oeste | CPAO, Dourados-MS | 25 à 29 de junho de 2012 |
| Capacitação em R | Conselho Nacional de Justiça | Brasília-DF | 20-12/06 e 04-05/10 |
| Modelagem e análise de dados experimentais com o programa computacional R | Pós Graduação em Produção Vegetal - UFPR | UFPR, Curitba-PR | 3º trimestre letivo de 2012 |
| Aplicação de modelos mistos por meio da ferramenta R | Pesquisadores da Embrapa Arroz e Feijão e acadêmicos da UFG | CNPAF, Santo Antônio de Goiás-GO | 12 à 14 de novembro de 2012 |
| Modelos de regressão não linear | Docentes do DCET da UESC | UESC, Ilhéus-BA | 22 à 23 de novembro de 2012 |
Blog
 Blog que administro e mantenho com a colaboração de amigos também apaixonados pelo R. Clique na figura ao lado para ir para o blog.
Blog que administro e mantenho com a colaboração de amigos também apaixonados pelo R. Clique na figura ao lado para ir para o blog.
Consultor
- Revista de Ciências Agrárias / Amazonian Journal of Agricultural and Environmental Sciences (revisor);
Artigos publicados em periódicos
- Consulte meu perfil acadêmico no Mendeley. Todas as publicações concluídas possuem cópia digital disponível para download.
Participação em Projetos/Grupos de Pesquisa
- Projeto com Larissa (http://lattes.cnpq.br/5306520242222948);
- Projeto com Maristella (http://lattes.cnpq.br/7578795880165011);
- Projeto com Eduardo (http://lattes.cnpq.br/0288146552740769);
- Projeto com Rafael (http://lattes.cnpq.br/3314759970388496);
- Projeto com Rodrigo (http://lattes.cnpq.br/3314759970388496);
- Grupo com PJ;
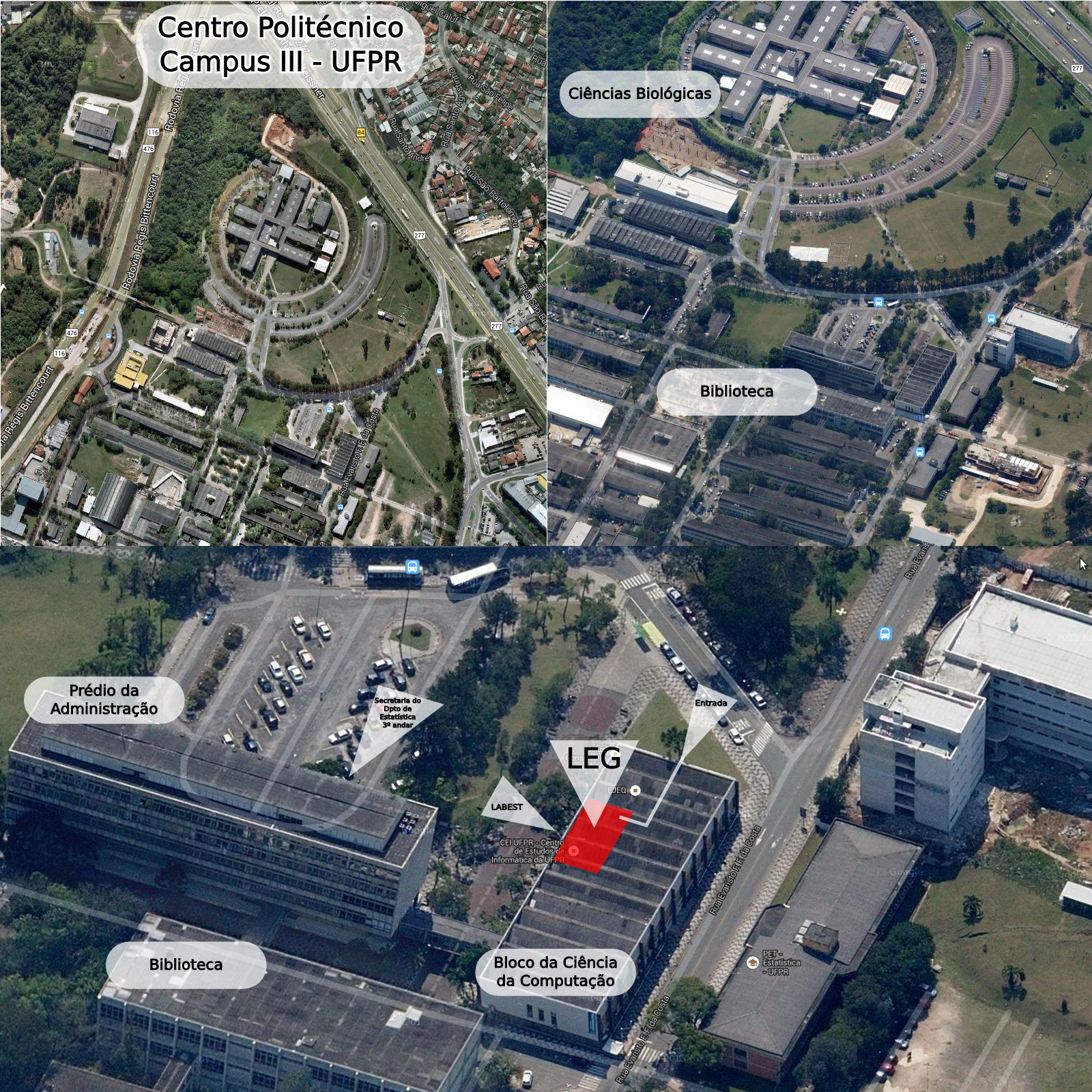
Localização e acesso ao LEG
O acesso ao LEG se dá pela escada/rampa localizada na face norte do bloco de Informática. Ao entrar pelo corredor do antigo salão de provas, procure por uma porta sinalizada do lado direito. Clique aqui para abrir a nossa localização em uma nova página com seta de indicação.
Dicas de Linux
- Estilo de citação ABNT para Mendeley;
- Estilos ABNT disponíveis: http://wisleyvelasco.com.br/2013/05/16/estilos-mendeley/
- Site para produzir expressões matemáticas em latex (http://www.codecogs.com/latex/eqneditor.php). Permite a construção online de expressões matemáticas em código Latex, que podem ser copiadas e coladas no seu editor. O usuário tem opção de usar os menus com expressões prédefinidas, tais como matrizes, sistema de equações, derivadas, somatórios, simbologia de conjuntos, letras gregas, etc. Expressões comuns estão organizadas no botão "exemplos".
- Download do LaTable para confecção de tabelas para Latex (http://www.leg.ufpr.br/~walmes/docs/LaTable.exe). A versão disponível para Download tem compatibilidade com o wine e portanto pode ser usada por usuários Linux (usando o wine). Nos casos em as tabelas estão prontas em editores de texto (e.g. OpenOffice-Writer) ou em planilhas eletrônicas (e.g. OpenOffice-Calc) pode-se colar o conteúdo da tabela em um arquivo de extensão *.txt e importar pelo LaTable. No LaTable é possível fazer a formatação de alinhamento de colunas e inserção linhas verticais e horizontais, usadas, em geral, para separar o cabeçalho da tabela do restante. Depois da edição é só copiar, em formato Latex, o conteúdo da tabela para área de transferência e colar no seu editor.
- Zotero: plugin do Firefox para extração de meta-dados de páginas de internet para criação de referências bibliográficas (http://www.zotero.org/).
Zotero é capaz de extrair os metadados de sites da internet e produzir referências bibliográficas em diversos formatos. Sites Amazon.com (de venda de livros) e o ScienceDirect (para busca de artigos) são alguns dos compatíveis com o plugin. Basta fazer a busca por um livro/artigo no site que um ícone irá aparecer na sua barra de endereços. Ao clicar no ícone (que será um livro de capa azul no caso de livros) os metadados serão organizados pelo zotero. Para acessar os metadados, clica-se no nome "zotero" que fica no canto inferior direito do seu Firefox. Pode-se exportar as referências dos livros/artigos (e outros tipos de publicação) para diversos formatos, inclusive para o formato usado pelo bibtex, além de formatos para OpenOffice-Writer, html, etc. Mais detalhes consultar a documentação no site do plugin. Consulte também: tutorial em pdf, descrição, video. - Site para criar senhas aleatórias, com diversas opções como tamanho e tipos de caracteres e criação de senhas pronunciáveis.
- Para converter as figuras em formato *pdf de um diretório para *jpg e fazer um zip delas no final (dica Linux). Salvar num arquivo com extensão *sh (e.g. converter.sh), dentro do diretório das figuras, o conteúdo abaixo (o tipo de aspas é importante)
for file in `ls *.pdf` do convert $file `echo $file | sed 's/\.pdf$/\.jpg/'` done zip todojpg.zip *.jpg rm *.jpgEntre no terminal do Linux. Vá para o diretório em que estão as figuras e o arquivo converter.sh (use $ cd <caminho>). Execute
$ chmod +x converter.sh $ ./converter.shNo final deverá haver um zip com as figuras em *jpg. Caso não queria que as figuras em *jpg sejam removidas do diretório, exclua a última linha arquivo *sh.
- Outra forma de fazer conversão é usando o ImageMagick, assim:
$ convert -density 300x300 -resize 1000x1000 *.pdf out.jpg
- para fazer com figuras tendo determinado prefixo
for file in `ls prefixo*.pdf` do convert -density 300x300 -resize 1000x1000 $file $file.jpg done zip todojpg.zip *.jpg rm *.jpgEntre no terminal do Linux. Vá para o diretório em que estão as figuras e o arquivo converter.sh (use $ cd <caminho>). Execute
$ chmod +x converter.sh $ ./converter.shNo final deverá haver um zip com as figuras em *jpg. Caso não queria que as figuras em *jpg sejam removidas do diretório, exclua a última linha arquivo *sh.
- Para fazer uma busca de uma expressão reugular dentro de uma árvore de diretórios apenas nos arquivos de determinada extensão. No caso, abaixo está ilustrada a procura dos arquivos de extensão R que possui a palavras 'try'
$ grep -r --include=*.R 'try' *
- trocar texto dentro de um arquivo. O texto 'walmes' será substituido por 'zeviani' dentro do arquivo experII.R
$ perl -i -pe 's/walmes/zeviani/;' experII.R
- para redimensionar as fotos (*jpg, ou gráficos do R, rs) de um diretório, deixando as fotos com largura de 640 pixels e mantendo proporção original, e forçando a proporção 640x480.
$ mogrify -resize 640 *.jpg $ mogrify -resize 640×480! *.jpg $ mogrify -resize 25% *.jpg
- colocar alias no wiki. Logar na pata** via console
$ ssh -p <porta> <usuário>@<endereçomáquina> $ sudo vim /etc/apache2/conf.d/alias (use esc : wq para sair e salvar) $ sudo /etc/init.d/apache2 restart
- para instalar pacotes do R no linux que possuem dependências de elementos do sistema operacional, geralmente pacotes gráficos (rgl, playwith, gWidgetsRGtk2), use o comando
$ sudo apt-get build-dep r-cran-nomedopacote
- para procurar e substituir no emacs dê
alt+shift+5(ou alt+%), abre o minibuffer, escrevenome de buscaENTER, escreve onome de substituiçãoENTER, dêalt+%para avançar na tarefa,ypara confirmar substituição,npara não substituir. - para mudar o cursor de buffer no emacs use
ctrl+c+o. - para deixar o gedit com corretor ortográfico em português http://beaglenetwork.blogspot.com/2010/05/corretor-ortografico-em-portugues-para.html.
- para converter arquivos de texto da codoficação utf-8 para a iso-8859-15
#!/bin/bash
# esse bash faz a conversão de arquivos R em codificação utf-8 para iso-8859-1
# $1 é para fornecer um nome para ir no nome arquivos, usar então o que está abaixo
$1
for a in *.R;
do
echo "Convertendo $a em utf-8 para ${a%%.R}-$1.R em iso-8859-15 ..."
iconv -f utf-8 -t iso-8859-15 "${a}" -o "${a%%.R}-$1.R"
done
Entre no terminal do Linux. Vá para o diretório em que estão os arquivos e o arquivo utf2iso.sh (use $ cd <caminho>). Execute
$ chmod +x utf2iso.sh $ ./utf2iso.sh
- para montar pendrive com permissões de escrita
$ sudo mount -o remount,rw /media/HP\ v115w
- remove os "_" (underlines) do nome dos arquivos e subtitui por espaços " "
$ find -type f -exec rename 'y/\_/\ /' {} \;
- remove o espaço antes de .pdf nos arquivos
$ find -type f -exec rename 's/\ \./\./' {} \;
- para compilar Sweave especificando a codificação de caracteres
$ R CMD Sweave --encoding=utf-8 arquivo.Rnw
- para criar bash que tranfere arquivos diretório web da disciplina. Salvar o conteúdo em um arquivo de extensão
*.sh, no casosobe.sh, e colocar no diretório/bin
#!/bin/sh echo "Transferindo os arquivos necessários para a página da Disciplina, requer senha ..." scp -P aaaa $1 walmes@bbb.cc.ddd.ee:/home/walmes/public_html/ensino/$2 echo "Todos os procedimentos foram executados com sucesso!"
$1 é o nome do arquivo a ser transferido e $2 é o diretório da disciplina. Feito isso, para usar basta fazer
sobe.sh arquivo.pdf estatistica_basica_2012
- Para concatenar arquivos pdf, útil quando fazemos provas personalizadas.
# http://rafaelbotelho.com/2009/12/13/concatenando-arquivos-pdf-no-linux/ gs -q -sPAPERSIZE=a4 -dNOPAUSE -dBATCH -sDEVICE=pdfwrite -sOutputFile=saida.pdf *.pdf
pdfjoin arquivo1.pdf arquivo2.pdf ... aquivon.pdf pdfjoin *.pdf
h-r4xfp-7i8cv-495t6-36jby-bs4tu
- Matrizes com Sweave
\left[\begin{array}{c|c}
1 & c_2 \\ \hline
0 & 1 \\
0 & 3
\end{array}\right]
<<echo=false, results=hide>>=
require(xtable)
m <- matrix(1:9, 3, 3)
aux <- paste(rep("c", ncol(m)), collapse="")
@
$\left(\begin{array}{\Sexpr{aux}}
<<echo=false, results=tex>>=
print(xtable(m), only.contents=TRUE, hline.after=NULL,
include.rownames=FALSE, include.colnames=FALSE)
@
\end{array}\right)$
- Para fazer um for em nomes de arquivos e aplicar determinado comando
# lista os arquivos de final R, aplica for para cada um
# bn pega o basename (sem a extensão) e usa como primeiro argumento do comando transferiR
for file in `ls *.R`
do
bn="${file%.*}"
transferiR $bn cursoR/af722_2012
done
Playground
Faça o download do script abaixo aqui: script.R.
#=============================================================================
# Uso dos escores de uma análise de fatorial como variável dependente em um
# modelo linear de experimento
#=============================================================================
#-----------------------------------------------------------------------------
# lê os dados e remove NA
da <- read.table("http://www.leg.ufpr.br/~walmes/cursoR/cnpaf/mudas2.txt",
header=TRUE, sep=";")
str(da)
da <- da[complete.cases(da),]
da$trat <- factor(da$trat) # atualiza os níveis de trat
da$bloc <- factor(da$bloc) # passa bloc para fator
str(da)
#-----------------------------------------------------------------------------
# experimento com mudas de caju em função de categorias de subtrato, arranjado
# em blocos ao acaso com 20 plantas por unidade experimental. Foram observados
# os valores individuais de cada planta que sobreviveram (viab=1). Com isso o
# surge desbalanceamento à nível de plantas.
#-----------------------------------------------------------------------------
# análise de variância para alt segundo o modelo experimental
m0 <- aov(alt~bloc+trat+Error(bloc:trat), data=da)
summary(m0)
#-----------------------------------------------------------------------------
# análise para todas as demais
m00 <- lapply(as.list(da[,-c(1:4)]),
function(x){
m0 <- aov(x~bloc+trat+Error(bloc:trat), data=da)
m0
})
lapply(m00, summary) # trat não significativo para todas as variáveis
#-----------------------------------------------------------------------------
# rodando análise fatorial, dados padronizados
X <- scale(as.matrix(da[,5:14]))
str(X)
pairs(X)
cor(X)
ei <- eigen(cor(X))
cumsum(ei$values)/sum(ei$values)
#-----------------------------------------------------------------------------
# obtenção dos fatores
nfat <- 3
af <- factanal(X, factors=nfat,
scores="regression", rotation="varimax")
print(af$loadings, cutoff=0.5, sort=TRUE)
# fator 1 - do caule: altura, diâmetro, massa seca e fresca
# fator 2 - das raízes: massa seca e fresca
# fator 3 - das folhas: massa seca e fresca
# comunalidade, 1-variância específica
1-sort(af$uniqueness)
# escores
S <- af$scores
plot(S)
#-----------------------------------------------------------------------------
# analisar os scores do 1 ao 3 fator
m00 <- apply(S, 2,
function(x){
m0 <- aov(x~bloc+trat+Error(bloc:trat), data=da)
m0
})
lapply(m00, summary) # trat significativo para os Fatores 2 e 3
#=============================================================================
# manova aplicada aos dados de *parcela*
#=============================================================================
#-----------------------------------------------------------------------------
# calcula as médias por bloc:trat para todas as variáveis
require(plyr)
dam <- ddply(da[,5:14], .(trat=da$trat, bloc=da$bloc), mean)
str(dam)
#-----------------------------------------------------------------------------
# manova, quero a matriz de covariância livre dos efeitos fixos
ma0 <- aov(as.matrix(dam[,3:12])~bloc+trat, data=dam)
anova(ma0)
str(ma0)
#-----------------------------------------------------------------------------
# matriz de covariância condicional
R <- residuals(ma0)
str(R)
#-----------------------------------------------------------------------------
# covariância e correlação resídual
cov(R) # é viesada porque tem denominador n-1 que é maior que n-p
cor(R)
#-----------------------------------------------------------------------------
# análise fatorial aplicada aos resíduos
nfat <- 3
af <- factanal(scale(R), factors=nfat,
scores="Bartlett", rotation="varimax")
print(af$loadings, cutoff=0.5, sort=TRUE)
# fator 1 - ?sem sentido
# fator 2 - ?sem sentido
# fator 3 - ?sem sentido
# comunalidade, 1-variância específica
1-sort(af$uniqueness)
# escores
S <- af$scores
plot(S)
#-----------------------------------------------------------------------------
# analisar os scores do 1 ao 3 fator
m00 <- apply(S, 2,
function(x){
m0 <- aov(x~bloc+trat, data=dam)
m0
})
lapply(m00, summary) # trat significativo para os Fatores 2 e 3
# não tem efeito porque eu removi o efeito antes da análise fatorial
# então explorei a relação entre as variâncias condicionais, que são
# livres do efeitos fixos.
# teria como eu obter os escores dos dados originais, baseados na
# covariância dos resíduos?
#-----------------------------------------------------------------------------
# devolvendo o efeito dos fatores fixos
# usando o preditor de mínimos quadrados, Daniel pg 527, equação 11.7.2
Psi <- cov(R)
Gamma <- t(af$loadings[,1:nfat])
F <- solve(Gamma%*%solve(Psi)%*%t(Gamma))%*%(Gamma%*%solve(Psi)%*%t(dam[,3:12]))
F <- t(F)
m00 <- apply(F, 2,
function(x){
m0 <- aov(x~bloc+trat, data=dam)
m0
})
lapply(m00, summary) # trat significativo para os Fatores 2 e 3
#-----------------------------------------------------------------------------
Playground 2
#----------------------------------------------------------------------------- # Simulando variáveis aleatórias #----------------------------------------------------------------------------- # EXERCÍCIO 1 - Considere as funções abaixo: # 4) f(x) = 2*sqrt(1-x^2)/pi, I(-1 < x < 1) # Com as funções curve() e integrate() do R, obtenha: # a) crie uma função R que calcule f(x); fx=function(x) 2*sqrt(1-x^2)/pi # b) verifique se a função é uma função densidade de probabilidade; # Para que f(x) seja uma fdp, é necessário que: # i) f(x) >= 0, I(-1 < x < 1) # Analisando a função f(x), verifica-se que quando -1 < x < 1, sqrt(1-x^2) será sempre # maior ou igual a zero. # ii) int f(x) dx = 1, I(-1 < x < 1) # Integrando a função f(x), temos: integrate(fx,-1,1) # portanto, f(x) é uma fdp. # c) faça o gráfico da função, tenha atenção quanto ao domínio da função; curve(fx,-1,1,ylab='f(x)',main='Gráfico da função densidade de x') # d) obtenha a média (valor esperado) dessa distribuição; # A média dessa função pode ser dada pela esperança matemática para v.a. contínua, dada por: # E(x) = int x f(x) dx # logo: Ex = function(x) x * 2*sqrt(1-x^2)/pi integrate(Ex,-1,1) # e) escolha dois pontos dentro do domínio da função e calcule a área delimitada, ou seja, # P(xi < X < xs); # Vamos calcular P(0 < X < 0.5) integrate(fx,0,0.5) polygon(c(0,seq(0,0.5,0.001),0.5),c(0,fx(seq(0,0.5,0.001)),0),col='lightgreen') # f) crie uma função R que calcule a F(x), ou seja, a função de distribuição acumulada; fa = function(x) sapply(x, function(xi) integrate(fx,-1,xi)$value) fa(c(0,1)) curve(fa,-1,1,ylab='f(x)',main='Gráfico da função densidade de x') # outra opção seria integrar fx, ou seja, # facum <- function(x) integrate(((2/pi)*(x/2*sqrt(1-x^2)+ 1/2*asin(x))), -1, x) # facum representa melhor a F(x) do que fa, pois é uma integração numérica # g) crie uma função R que calcule a F(x)-1, ou seja, a inversa da função de distribuição # acumulada; # Calcular a inversa da função de distribuição acumulada fa é bastante complicado, assim, # como alternativa, se plotarmos vários # valores para x com a fa, podemos interpolar esses pontos de forma a obtermos uma # aproximação para F(x)-1. x <- seq(-1, 1, length=100) y <- fa(x) plot(y~x) plot(x~y) ifa <- approxfun(x=y, y=x) # função que liga cada ponto xi por meio de uma função linear. curve(ifa, 0, 1, add=TRUE, col=2) # h) pelo método da transformação integral da probabilidade, gere 1000 números aleatórios # da distribuição f(x); u <- runif(1000) ax <- ifa(u) hist(ax, prob=TRUE, col='lightgreen') curve(fx, add=TRUE, col= 2) # Que legal!!! =D
Tikz lista de links
Lista com links sobre tikz