O experimento fatorial descrito em Banzato & kronka (1989) comparou o crescimento de mudas de eucalipto considerando como fatores diferentes tipos de recipientes e espécies.
Vamos considerar agora que os dados já estejam digitados em um arquivo texto. Clique aqui para ver e/ou copiar o arquivo com conjunto de dados para o seu diretório de trabalho. A seguir deve-se ler ("importar") os dados para R com o comando read.table(): Se voce não tiver restrições de acesso (firewall, etc) pode importar o arquivo diretamente fornecendo a URL (endereço web) do arquivo.
Antes de começar as análise vamos usar alguns comandos para inspecionar o objeto que contém os dados para saber quantas observações e variáveis há no arquivo, bem como o nome das variáveis. Vamos também pedir o R que exiba um rápido resumo dos dados e verificar se cada variável possui o "tipo"correto.
Nos resultados acima vemos que o objeto ex04 que contém os dados tem 24 linhas (observações) e 3 colunas (variáveis). As variáveis tem nomes rec, esp e resp, sendo que as duas primeiras são fatores enquanto resp é uma variável numérica, que no caso deste experimento é a variável resposta.
Inicialmente vamos obter um resumo de nosso conjunto de dados usando a função summary(). Note que para os fatores são exibidos o número de dados em cada nível do fator. Já para a variável numérica são mostrados algumas medidas estatísticas.
Vamos explorar um pouco mais os dados calculando as médias para cada nível de cada fator e também para as combinações dos nívies dos fatores.
As combinações dos níveis dos fatores podem ainda ser obtidas com interaction() que produz uma saída na forma de um vetor com nomes que combinam os níveis dos fatores envolvidos.
Nos comandos mostrados anteriormente a função mean() pode ser substituída por qualquer outra função de interesse seja pré definida ou definida pelo usuário. Nos exemplos a seguir ilustramos ambas situações onde são obtidas as medianas com a função pré-definida mediam e o número de observações acima de 22 para cada combinação dos fatores, com uma função definida por nós.
As médias para so fatores e suas combinações também poderiam ser obtidas com o comando model.tables() o que será mostrado mais adiante. Entretanto neste estágio de análise descritiva, preferimos o mecanismo mais geral de tapply() que permite o cálculo de outros resumos além da média. Experimente nos comandos acima substituir mean por var para calcular a variância de cada grupo, e por summary para obter um outro resumo dos dados.
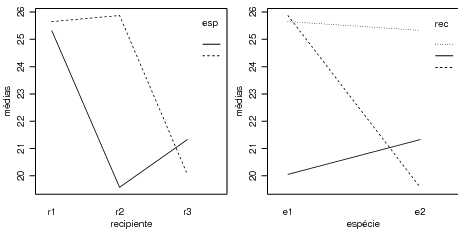
Em experimentos fatoriais é importante verificar se existe interação entre os fatores. Inicialmente vamos fazer isto graficamente e mais a frente faremos um teste formal para presença de interação. Os comandos a seguir são usados para produzir os gráficos exibidos na Figura 25.2.
Pode-se usar o R para obter outros tipos de gráficos de acordo com o interesse de quem está analisando os dados. Os comandos a seguir ilustram alguns outros tipos de gráficos que podemos produzir. Na figura 25.2 são mostrados gráficos semelhantes aos mostrados anteriormente, porém com pontos referentes às observações o que permite visualizar a variabilidade em cada grupo definido pelas combinações dos níveis dos fatores.
Além destes gráficos produzidos pelo sitema básico de gráficos do R pode-se usar comandos fornecidos pelo pacote lattice que implementam um poderoso conjunto alternativo de gráficos mas não serão abordados aqui.
Seguindo o modelo adequado, o análise de variância para este experimento inteiramente casualizado em esquema fatorial pode ser obtida com as funções aov() ("analysis of variance") ou lm() ("linear model"). A primeira usa a segunda internamente visto que o modelo é linear, porém ajusta os resultados em um formato em geral mais adequado para análise de experimentos. Nestas funções os modelos são declarados por "fórmulas". A seguir vemos duas fórmulas que especificam o mesmo modelo.
Isto significa que dentro de uma fórmula no R, o símbolo ":" define o termo de interação e "*" indica da inclusão dos efeitos principais e interações. A análise acima mostra que neste caso o efeito de interação é significativo, confirmando o que for indicado nos gráficos exploratórios do efeito de interação vistos anteriormente.
O objeto ex04.av guarda todos os resultados da análise e pode ser explorado por diversos comandos. Por exemplo a função model.tables aplicada a este objeto da classe aov produz tabelas dos efeitos (se type="effects") ou das médias (se type="effects") definidas pelo modelo. O resultado mostra a média geral, médias de cada nível dos fatores e das combinações dos níveis dos fatores. No resultado está incluído também o número de dados que gerou cada média.
Mas isto ainda não é tudo que se pode extrair da análise! O objeto ex04.av possui vários elementos que guardam diversas outras informações sobre o ajuste do modelo e que podem ser exploradas subsequentemente por métodos de funções para as classes aov e lm ou por requisições definidas pelo usuário. A seguir veremos alguns exemplos.
A chamada class() mostra que o objeto ex04.av pertence às classes aov e lm. Isto significa que devem haver métodos associados a este objeto que tornam a exploração do resultado mais fácil. Na verdade já usamos este fato acima quando digitamos o comando summary(ex04.av). Existe uma função chamada summary.aov() que foi utilizada já que o objeto é da classe aov. Iremos usar mais este mecanismo no próximo passo da análise, a análise de residuos.
A análise de resíduos é útil para verificar os pressupostos do modelo. Usando o mecanismos de classes, o comando plot(ex04.av) aplicado sobre o objeto que contém o ajuste do modelo produz uma figura com quatro gráficos básicos para análise dos resíduos conforme mostrado na Figura 25.4.
Os gráficos permitem uma análise dos resíduos que auxilia no julgamento da adequacidade do modelo. Evidentemente não é necessario limitar-se aos gráficos produzidos automaticamente pelo R – voce pode criar os seus próprios gráficos. Neste gráficos pode-se usar outras variáveis, tipos de gráficos, mudar texto de eixos e títulos, etc, etc, etc. Os comandos a seguir mostram como obter os gráficos boxplot dos resíduos para os níveis de cada um dos fatores como mostrado na Figura 25.4.
A Figura 25.4 mostra outros gráficos definidos pelo usuário: resíduos versus valores preditos, um boxplot dos resíduos padronizados, e um qqplot dos resíduos do modelo. Note que o objeto que contém o ajuste foi utilizado para extrair resíduos, valores preditos e a estimativa s2 da variância dos resíduos.
Além da análise gráfica de resíduos há alguns testes já programados em funções. Como exemplo vejamos o teste de Shapiro-Wilks para testar a normalidade dos resíduos.
Quando a interação entre os fatores é significativa pode-se adotar como estratégia de análise o desdobramento dos graus de liberdade de um fator dentro de cada nível do outro fator. Uma forma de obter tal desdobramento no R é reajustar o modelo utilizando a notação / que indica efeitos aninhados. Desta forma podemos desdobrar os efeitos de espécie dentro de cada recipiente e vice versa conforme mostrado a seguir.
Neste ponto vale uma explicação mais detalhada sobre como obter os desdobramentos da interação usando o argumento split, em particular como definir os elementos da lista que, no caso de ex04.ave foram e1=c(1,3) e e2=c(2,4). Iniciamente vamos extrair usando effects() os efeitos ajustados pelo modelo.
Os efeitos que temos interesse no desdobramento são os da interação, que são: espe1:recr2, espe2:recr2, espe1:recr3 e espe2:recr3. Portanto temos que localizar no vetor de efeitos as posições desses efeitos de interação que são: 1o : espe1:recr2, 2o : espe2:recr2, 3o : espe1:recr3 e 4o : espe2:recr3. Isto mostra que a posição dos efeitos que contém a espécie1 (e1) são 1 e 3, e especie2 (e2) são 2 e 4 o que define os valores nos vetores indicados no argumento split.
Há vários testes de comparações múltiplas disponíveis na literatura, e muitos deles são implementados nos pacotes básicos do R e/ou em pacotes contribuídos. Por exemplo, o pacote multcomp é inteiramente dedicado à implementação de diversos procedimentos de comparações múltiplas no R. Além disto, procedimentos que não estejam implementados podem ser calculados utilizando os recursos usuais do R utilizando os objetos com o ajuste dos modelos. Como ilustração mostramos a seguir duas formas de obter resultados para o Teste de Tukey, a primeira usando uma implementação já disponível com a função TukeyHSD() e uma segunda sem fazendo os cálculos necessários passo a passo com operações básicas do R. Para função já disponível simplesmente digitamos os comandos a seguir e os resultados podem ser mostrados na forma texto ou gráfica como na Figura 25.6 que é produzida com o comando plot(ex04.tk1).
Esta saída fornece resultados detalhados de várias comparações possíveis entre os níveis dos fatores e suas combinações. Entretanto, neste caso, nem todos os resultados mostrados nos interessam. Como a interação foi significativa na análise deste experimento a comparação dos níveis fatores principais não nos interessa. Podemos então pedir a função que somente mostre a comparação de médias entre as combinações dos níveis dos fatores e o gráfico com tais resultados pode ser obtido com plot(ex04.tk2).
Mas ainda assim temos resultados que podem não interessar. Mais especificamente, considere que estamos intessados nas comparações dos níveis de um fator dentro de cada um dos níveis do outro fator. Neste ponto, vamos fazer as comparações dos recipientes para cada uma das espécies, fazendo os cálculos passo a passo. Primeiro vamos obter a estimativa da variância dos resíduos, que é usada junto com o valor da amplitude estudantizada fornecida por qtukey() para obter o valor da diferença mínima significativa que no código a seguir armazenamos no objeto dt.
Este valor é então usado para comparar as médias de interesse. Anteriormente armazenamos as médias para as combinações de todos os níveis dos fatores no objeto ex04.m onde as linhas se referem aos recipientes e colunas às espécies. No objeto m1 armazenamos as médias para espécie1 e na sequência são feitos cálculos para verificar a significância da diferença entre as médias dos recipientes para esta espécie.
No código mostrado anteriormente fazemos alguma manipulação dos objetos para formatar a saída. Esta sequência pode ser usada para definir uma função o que evitaria a digitação de todos estes comandos a cada comparação de médias desejada. Procedimento análogo pode ser adotado para fazer outras comparações de interesse.