Nesta sessão iremos usar o R para analisar um experimento em delineamento inteiramente casualizado com apenas um fator. Tal procedimento é também chamado em alguns textos de "análise da variância de simples entrada"(one-way anova). A seguir são apresentados os comandos exemplificando alguns procedimentos usuais para a análise dos dados de um experimento deste tipo que, neste exemplo, envolve um fator com nove níveis (tratamentos). O primeiro passo é ler os dados.
Caso não consiga executar o comando acima diretamente com o endereço http utilize um navegador para ir até esta página e copie o arquivo exemplo1.txt para o seu diretório de trabalho. Caso o arquivo esteja em outro diretório deve-se colocar o caminho completo deste diretório no argumento de read.table() acima. A seguir vamos inspecionar o objeto que armazena os dados e seus componentes. Em particular é importante certificar-se que a variável resposta é do tipo numeric e, se os níveis de tratamentos forem qualitativos, a variável indicadora dos tratamentos é do tipo factor . Caso isto não ocorra é necessário transformar as variáveis para estes tipos antes de prosseguir com as análises.
Portando o objeto ex01 é um data-frame com duas variáveis, sendo uma delas um fator (a variável trat) e a outra uma variável numérica (resp). Vamos iniciar obtendo um rápido resumo dos dados que mostra que este é um experimento "balanceado"com mesmo número de repetições (seis) para cada tratamento. Calculamos também as médias, variâncias e erros padrão das médias para cada tratamento separadamente.
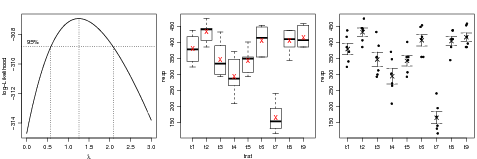
Vamos prosseguir com a análise exploratória com gráficos gerados pelos comandos a seguir e mostrados na Figura 24. O gráfico de esquerda utiliza a função boxcox() do pacote MASS para verificar a necessidade de transformação dos dados o que neste caso não é necessária visto que o valor um está contido no intervalo definido pelas lines tracejadas. A transformação Box-Cox é discutida me mais detalhes em uma outra Seção deste material. O gráfico do meio mostra um boxplot para os dados de cada tratamento, o que deve ser analisado com cautela lembrando que cada boxplot é produzido com apenas seis observações. Optamos aqui por indicar também neste gráfico a média de cada tratamento. O gráfico da direita produzido com stripchart() é uma alternativa ao boxplot para amostras de tamanho pequeno. Na chamada desta função optamos por alterar valores default de alguns argumentos como por exemplo para method="jitter" que provoca pequeno um deslocamento horizontal aleatório dos pontos evitando assim sobreposição de pontos com valores coincidentes ou muito próximos. Ainda neste gráfico acrescentamos as médias e barras que somam e subtraem os erros padrões da média para cada tratamento. Na função arrows() os quatro argumentos iniciais informam coordenadas para as barras, code=3 informa que as "setas"devem ser colocadas em ambas extremidades e angle=90 faz com que a "seta"se torne uma pequena barra horizontal com o tamanho controlado por length.

É importante notar que as barras simplesmente refletem a variância dos dados dentro da cada tratamento e não são adequadas para detectar diferenças entre tratamentos, o que será discutido mais adiante nesta sessão. Além dos gráficos acima podemos também verificar o pressuposto de homogeneidade de variâncias com o testes de igualdeda de variâncias, como por exemplo, o teste de Bartlett. Neste caso o teste indica variâncias homogêneas. Caso isto não ocorresse uma possível alternativa seria usar o procedimento descrito na Sessão 24.3.
Uma vez concluída a análise exploratória e verificada a adequacidade de alguns pressupostos o passo seguinte é ajustar o modelo usando aov() ou lm(). Neste exemplo, por se tratar da análise de um experimento, tipicamente avaliada pelo quadro de análise de variância, optamos por usar aov(). Embora aov() use lm() internamente, os resultados são oranizados internamente de forma conveniente para a efetuar a análise de variância.
Portanto o objeto ex01.mod é uma lista que guarda os resultados da análise para o modelo ajustado. Vamos inspecionar este objeto e seus elementos mais detalhadamente ilustrando como usá-lo para obter a análise dos resultados e extrair elementos para a análise de resíduos. A função names() mostra os elementos da lista e adicionalmente existem funções que extraem elementos do objeto. Duas tipicamente utilizadas são coef() para extrair os coeficientes, residuals() para extrair resíduos e fitted() para valores ajustados, mas há ainda várias outras como effects(), AIC() logLik(), model.tables(), entre outras.
O resultado de coef() vai depender da parametrização adotada e definida pelos contrastes. Os valores default e/ou correntes são dados por options()$contrasts. Para fatores qualitativos como no caso deste exemplo a parametrização default corresponde a "contr.treatment" que assinala o valor da média do primeiro tratamento (primeiro nível do fator) ao primeiro coeficiente. Os demais representam a diferença das médias de cada um dos tratamentos à este tratamento de referência.
Uma outra forma de expecificar o modelo para este exemplo é mostrada a seguir com o uso -1 que, para níveis quantititivos corresponde a ajustar um modelo com intercepto igual a zero. No caso de níveis qualitativos como neste exemplo, monta uma matrix do modelo de forma a que cada coeficiente corresponda à média de cada um dos tratamentos. Note que apenas a interpretação dos coeficientes muda e a análise de variância permanece a mesma.
A parametrização para os coeficientes é determinada pela matriz do modelo e é definida pelo argumento contrasts de options() ou pela função contrasts() que mostra ou atribui a matrix de contrastes a ser utilizada. Fatores são definidos como sendo unordered (por exemplo nívies qualitativos como no caso da análise vista aqui) ou ordered, o que é usado, por exemplo, no caso de níveis quantitativos.
Para definir a parametrização a ser utilizada e definida pelos contrastes, pode-se usar outras opções de contrastes já disponibilizadas pelo R tipicamente usando options(). Nos comandos a seguir alteramos a opção para fatores unordered para "contr.sum". Os coeficientes obtidos são diferentes dos obtidos anteriormente sendo o primeiro a média geral e os demais uma comparação da média de cada tratamento contra as médias dos demais. Os resultados da análise de variância permanecem inalterado.
Os contrastes já definidos no R são listados e descritos a seguir. Além destes outros pacotes podem ter outras definições de contrastes, como em de "contr.sdif" do pacotes MASS. Estes contrastes são terão efeito se o termo -1 não for incluído no modelo pois neste caso os coeficientes são sempre as médias de cada um dos tratamentos, independente da opção de contraste adotada.
Além dos contrastes pré definidos, outros contrastes definidos pelo usuário e atribuídos ao fator em estudo usando a função contrasts(). Retornamos a este tópico com um exemplo na Sessão 24.1.3.
Retornando à análise do exemplo, vaos ver agora alguns gráficos e ferramentas para avaliar o modelo ajustado. Um método associado a plot() produz automaticamente gráficos de resíduos para objetos das classes lm e aov conforme ilustrado na Figura 24 produzida com o comando plot(ex01.mod).
Além dos gráficos "pré-preparados"pelo R, o usuário pode obter outros que desejar extraindo a informação necessária do objeto que contém o ajuste do modelo. Na Figura 24 mostramos quatro gráficos: resíduos padronizados versus valores preditos, boxplot, histograma dos resíduos padronizados e qqplot() dos resíduos. Para isto obtemos os resíduos padronizados dividindo os resíduos do modelo pela raiz quadrada da variância do termo de erro.
Um teste de normalidade dos residuos pode ser efetuado como indicado a seguir.
Uma das formas possíveis formas de interpretar os resultados no caso de efeito de tratamentos significativos é utilizar algum procedimento de comparações de tratamentos após verificar o resultado da anova, o que justifica o termo às vezes utilizado que descreve tais procedimentos como comparações post-hoc.
A questão do uso de comparações de tratamentos é polêmica no meio estatístico e não vamos aqui entrar em tal discussâo. Neste sessão vamos ilustrar três procedimentos deixando a cargo do leitor o julgamento de qual procedimento é mais adequado para o problema em questão. Os procedimentos discutidos a seguir correspondem a três possiveis abordagens ao problema de comparação de mais de duas médias, sendo eles: (i) teste-t para comparações duas a duas, (ii) teste de Tukey e (iii) contrastes e contrastes ortogonais. O primeiro caso se desdobra em mais opções uma vez que permite que os valores p sejam ou não ajustados, e caso sejam, por diferentes métodos.
Os procedimentos mostrados aqui são implementados em pacotes básicos do R. O pacote multcomp disponibiliza uma extensa lista de procedimentos adicionais de comparações múltiplas e alguns procedimentos específicos podem ainda ser encontrados em outros pacotes do R.
A função pairwise.t.test() calcula todas as possíveis comparações entre dois grupos, podendo ser vista como uma extensão ao teste-t para duas amostras, retornando o valor-p para cada comparação. A principal diferença é que o nível de significância deve ser corrigido para garantir o nivel de significância conjunto para todas comparações. O argumento p.adjust.method da função permite o usuário escolher entre diferentes métodos propostos para ajustar o nível de significância sendo o default o prodedimento proposto por Holm, que é uma modificação ao ajuste de Bonferroni, que também é disponível utilizando através do argumento p.adj="bonferroni". Mais detalhes podem ser encontrados na documentação da função.
O teste Tukey de comparações múltiplas é implementado na função TukeyHSD(). A saída em formato texto do teste de Tukey é mostrada a seguir e plot(ex01.HSD) produz o gráfico mostrado na Figura 24.1.2. As saídas da função mostram intervalos de confiança para as diferenças entre pares de médias.
Visualizações mais convenientes dos resultados podem ser obtidas com operações sobre o objeto resultante, tal como a usualmente adotada de listar as médias em ordem descrescente e indicar com letras as diferenças significativas ou não entre estas médias. Vamos ilustrar aqui uma possivel forma de obter tal visualização. Inicialmente vamos obter a DMS (diferença mínima significativa). No caso deste experimento balanceado, isto é, o mesmo número de repetições em cada tratamento, o intervalo de confiança para cada diferença é o mesmo e a DMS é portanto comum e dada por metade da amplitude do intervalo.
O passso seguinte é ordenar as médias deforma decrescente e verificar as diferenças significativas. O código abaixo é uma (mas certamente não a única) maneira de indicar as diferenças significativas código de letras usual na literatura.
Neste caso o procedimento é simples pois para um experimento balanceado e pelo teste de Tukey tem-se apenas um único valor de DMS. O algorítmo deve ser modificado e generalizado para outras situações ou pode-se usar funções de pacotes como multcompLatters do pacote multcompView.
Na análise de experimentos pode-se ter interesse em estudar determinadas comparações entre as médias que podem ser especificadas pelo usuário na forma de contrastes, que são um caso particular das funções estimáveis para o modelo. Vamos iniciar revendo definições.
Seja o modelo linear escrito na forma matricial Y = Xβ + ϵ onde Y é a variável resposta, X a matrix do modelo, β o vetor de p parâmetros (coeficientes) e ϵ o vetor de erros. Uma combinação linear dos coeficientes da forma ∑ pλpβp onde λ = [λ1,…,λp] é um vetor de constantes é dita uma função estimável para o dado modelo se λ pode ser escrita como uma combinação linear das linhas da X. Um contraste é um caso especial de função estimável em que a soma das constantes é nula, isto é, pode ser escrito como ∑ pcpβp onde ∑ pcp = 0.
No que se segue vamos ver como obter estimativas de contrastes de interesse no R, onde fórmulas lineares são usadas para definir as matrizes do modelo usadas no ajuste de modelos lineares e lineares generalizados. No caso de fatores (qualitativos) a matriz X do modelo não é definida unicamente para um mesmo experimento, podendo ser escrita de diversas formas alternativas que irão produzir a ajustes equivalentes. Tais formas são definidas pela escolha de contrastes ou funções estimáveis que definirão a interpretação dos coeficientes β do modelo. Portanto, se o interesse é apenas na análise de variância a particular forma adotada é irrelevante. Por outro lado, a escolha deve ser adequada se os coeficientes devem ser interpretados.
Ao ajustar um modelo as estimativas de contrastes podem ser obtidas de duas formas:
Vamos discutir aqui algums idéias iniciais sobre como implementar a segunda forma. Como na análise de contrastes os coeficientes passam a ser diretamente interpretados, passamos a usar lm() no ajuste do modelo.
Uma classe especial de contrastes é a de contrastes ortogonais. Um conjunto de contrastes ortogonais tem a propriedade de que as soma dos produtos dos coeficientes de qualquer par de contrastes deste conjunto é nula. Contrastes ortogonais são particularmente interessantes pois permitem desdobrar (particionar) a soma de quadrados de tratamentos um parcelas referentes a cada um dos contrastes. Isto permite que cada contraste seja testado diretamente por um teste t (ou o equivalente teste F).
Com nove tratamentos é possível definir oito contrastes ortogonais com cada um deles sendo associado a um dos graus de liberdade dos tratamentos. A definição destes contrastes não é única e deve refletir comparações relevantes para o problema em questão, assegurando-se que a ortogonalidade seja mantida o que garante que a soma das somas de quadrados dos contrastes seja equivalente à soma de quadrados total dos tratamentos. Para obter o desdobramento abordamos a modelagem como um problema de regressão múltipla onde os contrastes definem variáveis quantitativas a serem incluídas no modelo que é ajustado com lm(). Neste exemplo vamos considerar o seguinte conjunto de contrastes entre as médias dos tratamentos que são especificados nas linhas de uma matriz como se segue.
O próximo passo é fazer com que a matriz do modelo seja montada pelo R de forma que os coeficientes reflitam os contrastes desejados. Para isto associamos ao fator que representa os tratamentos (trat no exemplo) o atributo contrast contendo a inversa generalizada obtida por ginv() do pacote MASS. A analise de variância deste modelo é a mesma obtida anteriormente. entretanto os coeficientes são agora dados pela média geral seguida pelas estimativas de cada um dos oito contrastes definidos que que podem ser testadas diretamente pelo teste-t usando o comando summary().
Nos comandos a seguir visualizamos os mesmos resultados de uma forma alternativa, usando model.matrix() para montar a matrix de covariáveis da forma desejada, onde excluímos o intercepto (primeira coluna) e, para visualização adequadoa dos resultados, trocamos os nomes das colunas. A este data-frame adicionamos os dados e ajustamos o modelo de regressão com lm(). A função anova() sobre o modelo ajustado exibe a soma de quadrados decomposta entre os contrastes agora testados pelo teste F que é equivalente ao teste-t mostrado acima pois cada contraste possui um grau de liberdade. Note que a soma delas corresponde a soma de quadrados de tratamentos mostrada no ajuste inicial do modelo o os coeficientes são os mesmos.
Os coeficiente retornados equivalem à aplicar os contrastes desejados sobre as médias dos tratamentos. Pode-se ainda visualizar os contrastes assinalados ao fator trat através da inversa generalizada.
Uma forma alternativa talvez mais direta e conveniente de obter a decomposição da soma de quadrados de tratamentos entre os contrastes é com o uso de aov() e summary() utilizando o argumento split
Nota: A atribuição do atributo contrast ao fator não terá efeito sobre a construção da matrix do modelo caso o termo de intercepto esteja retirado na definição do modelo, por exemplo, se o modelo acima fosse definido por resp trat - 1.
Para cancelar a atribuição dos contrastes a um fator e retornar a definida por option() basta fazer atribuir a valor NULL.
Interpretando os contrastes: a interpretação dos contrastes vai depender dos valores dos coeficientes definidos na matriz do contrastes (matriz c1 no exemplo). Por exemplo suponha que desejamos obter contrastes de forma não só a obter testes de significância mas também obter estimativas dos contrastes com interpretação desejada. No exemplo, para comparar médias μi (ou efeitos ti) dos tratamentos com efeitos estimados do contrastes na mesma escala da médias do tratamentos poderíamos definir c1 da forma a seguir. Todos os resultados e testes de significância (estatístics de testes e p-valores) permanecem inalterados e apenas os valores dos efeitos estimados são diferentes
Definindo contrastes com funções do pacote gmodels . Este pacote oferece várias funções auxiliares que facilitam o uso e a apresentação dos resultados em formatos por vezes convenientes. No exemplo a seguir vamos repetir a obtenção dos resultados para os contrastes definidos ma matrix c1 conforme definida anteriormente.
Experimentos desbalanceados: finalmente vale ressaltar que o exemplo acima tratou de um experimento balanceado, isto é, com o mesmo número de repetições para cada tratamento e no caso de desbalanceamento ajustes são necessários na definição dos contrastes.
Na sessão anterior discutimos a comparação post-hoc de tratmentos utilizando funções como pairwise.t.text() e TukeyHSD implementadas no conjunto de pacotes básicos do R.
Outros procedimentos sãqo implementados em pacotes contribuídos do R. Entre estes encontra-se os pacotes multcomp e multcompView que implementam diversos outros procedimentos e gráficos para visualizações dos resultados. Vale notar que estes pacotes devem ser instalados com a opção dependencies=TRUE para garantir plena funcionalidade pois suas funções dependem de diversos outros pacotes.
Para ilustrar o uso desta pacote vamos efetuar novamente o teste de Tukey visto acima porém agora utilizando cálculos e gráficos gerados por funções destes pacotes, cujos resultados, embora iguais, são apresentados em forma diferente do visto anteriormente. A indicação de letras para diferenças entre pares de tratamentos mostrada a seguir requer que TukeyHSD seja invocada sem a ordenação dos tratamentos e uma representação visual é dada na Figura 24.2.
No caso de variâncias não homogêneas em experimentos inteiramente casualizados a função oneway.test() pode ser utilizada nas análises. Uma outra alternativa é a análise não paramétrica da Kruskall-Wallis implementada por kruskal.test().