1 Uma primeira sessão com o R
Esta é uma primeira sessão com o R visando dar aos participantes uma idéia geral da aparência e
forma de operação do programa. Os comandos abaixo motivam explicações sobre características
básicas de linguagem e serão reproduzidos, comentados e discutidos com os participantes durante o
curso.
Vamos começar gerando dois vetores x e y de coordenadas geradas a partir de números
pseudo-aleatórios e depois inspecionar os valores gerados.
[1] -1.1584296 2.0265046 -0.9943093 1.2948114 0.9499178
[1] -1.1584296 2.0265046 -0.9943093 1.2948114 0.9499178
[1] -1.158 2.027 -0.994 1.295 0.950
[1] 1.2299913 -0.9408620 0.9974967 -0.7250306 0.4638293
function (n, mean = 0, sd = 1)
NULL
No exemplo acima primeiro geramos um vetor x com 5 elementos. Note que ao fazermos y <- rnorm(x)
nao especificamos o tamanho da amostra explicitamente como anteriormente mas estamos definindo
um vetor y que tem o mesmo tamanho de x, por isto y foi gerado com também 5 elementos. Note que
se voce tentar reproduzir este exemplo deve obter valores simulados diferentes dos mostrados
aqui.
Ao digitar o nome do objeto x os elementos deste objetos são exibidos. O comando print(x)
também exibe os elementos do objeto porém é mais flexível pois oferece opções extras de
visualização. O comando print(x, dig=3) exibe este particular objeto x com no mínimo 3 dígitos
significativos. Para controlar o número de dígitos globalmente, isto é, para impressão de qualquer
objeto, por exemplo com 4 dígitos, usamos options(digits=4).
Neste simples exemplo introduzimos várias idéias e conceitos: objeto, atribuição de valores, vetores,
impressão de objetos, função, argumentos de funções, "defaults", geração de números aleatórios e
controle de semente.
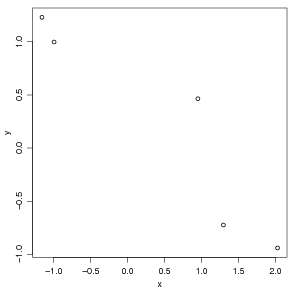
Agora vamos colocar num gráfico os pontos gerados usando o comando
Note que a janela gráfica se abrirá automaticamente e exibirá o gráfico. Há muitas opções de controle e
configuração da janela gráfica que são especidicadas usando-se a função par(). Algumas destas
opções serão vistas ao longo deste material.
A função plot() oferece através de seus argumentos várias opções para visualização dos gráficos.
As argumentos e básicos são mostrados a seguir.
function (x, y = NULL, type = "p", xlim = NULL, ylim = NULL,
log = "", main = NULL, sub = NULL, xlab = NULL, ylab = NULL,
ann = par("ann"), axes = TRUE, frame.plot = axes, panel.first = NULL,
panel.last = NULL, asp = NA, ...)
NULL
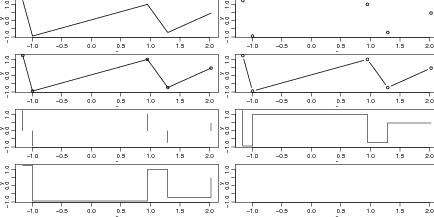
Para ilustração, no exemplo a seguir mostramos o uso do argumento type. Para facilitar esta
ilustração vamos primeiro ordenar os valores de x e y na sequência crescente dos valores de x.
> x <- sort(x)
> y <- y[order(x)]
Nos comandos abaixo iniciamos dividindo a janela gráfica em 8 partes e reduzindo as margens do
gráfico. A seguir produzimos diversos gráficos com diferentes opções para o argumento
type. Ao final retornamos a configuração original de apenas um gráfico na janela gráfica.
Um pouco mais sobre manipulação de vetores. Note que os colchetes [] são usados para selecionar
elementos e há funções para arredondar valores.
[1] -1.1584296 -0.9943093 0.9499178 1.2948114 2.0265046
[1] -0.9943093 0.9499178 1.2948114
[1] -1.2 -1.0 0.9 1.3 2.0
Os objetos existentes na área de trabalho pode ser listados usando a função ls() e objetos podem
ser removidos com a função rm(). Nos comandos a seguir estamos verificando os objetos
existentes na área de trabalho e removendo objetos que julgamos não mais necessários.
A seguir vamos criar um vetor que chamaremos de x com uma sequência de números de 1 a 20.
Depois criamos um vetor w de pesos com os desvios padrões de cada observação. Na sequência
montamos um data-frame de 3 colunas com variáveis que chamamos de x, y e w. Inspecionando o
conteúdo do objeto criado digitando o seu nome. A terminamos apagando objetos que não são mais
necessários.
[1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
[1] 1.500000 1.707107 1.866025 2.000000 2.118034 2.224745 2.322876 2.414214 2.500000
[10] 2.581139 2.658312 2.732051 2.802776 2.870829 2.936492 3.000000 3.061553 3.121320
[19] 3.179449 3.236068
> dummy <- data.frame(x = x, y = x + rnorm(x) * w, w = w)
> dummy
x y w
1 1 1.5134167 1.500000
2 2 0.5108889 1.707107
3 3 3.8646610 1.866025
4 4 2.9698978 2.000000
5 5 3.6280840 2.118034
6 6 5.1372141 2.224745
7 7 4.0745573 2.322876
8 8 8.3395924 2.414214
9 9 10.0991717 2.500000
10 10 8.5394195 2.581139
11 11 14.9127871 2.658312
12 12 12.8874653 2.732051
13 13 8.5623611 2.802776
14 14 13.0209883 2.870829
15 15 13.8633465 2.936492
16 16 16.5102130 3.000000
17 17 12.2013277 3.061553
18 18 23.6649292 3.121320
19 19 17.5954538 3.179449
20 20 12.7695068 3.236068
Nos comandos a seguir estamos ajustando uma regressão linear simples de y em x e examinando os
resultados. Na sequência, uma vez que temos valores dos pesos, podemos fazer uma regressão
ponderada e comparar os resultados.
> fm <- lm(y ~ x, data = dummy)
> summary(fm)
Call:
lm(formula = y ~ x, data = dummy)
Residuals:
Min 1Q Median 3Q Max
-5.6783 -1.1512 0.0336 1.1914 7.0518
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.1014 1.3423 0.076 0.941
x 0.9173 0.1121 8.186 1.76e-07 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 2.89 on 18 degrees of freedom
Multiple R-squared: 0.7883, Adjusted R-squared: 0.7765
F-statistic: 67.02 on 1 and 18 DF, p-value: 1.763e-07
> fm1 <- lm(y ~ x, data = dummy, weight = 1/w^2)
> summary(fm1)
Call:
lm(formula = y ~ x, data = dummy, weights = 1/w^2)
Residuals:
Min 1Q Median 3Q Max
-1.80255 -0.53011 -0.02346 0.56315 2.22021
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -0.07449 0.91136 -0.082 0.936
x 0.93386 0.09293 10.049 8.28e-09 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 1.007 on 18 degrees of freedom
Multiple R-squared: 0.8487, Adjusted R-squared: 0.8403
F-statistic: 101 on 1 and 18 DF, p-value: 8.28e-09
Gráficos de resíduos são produzidos com plot(). Como a função produz 4 gráficos dividiremos a
tela gráfica,
Note que o comando acima par(mfrow=c(2,2)) dividiu a janela gráfica em 4 partes para
acomodar os 4 gráficos. Para restaurar a configuração original usamos
Tornando visíveis as colunas do data-frame.
[1] ".GlobalEnv" "package:tools" "package:stats" "package:graphics"
[5] "package:grDevices" "package:utils" "package:datasets" "package:methods"
[9] "Autoloads" "package:base"
> attach(dummy)
> search()
[1] ".GlobalEnv" "dummy" "package:tools" "package:stats"
[5] "package:graphics" "package:grDevices" "package:utils" "package:datasets"
[9] "package:methods" "Autoloads" "package:base"
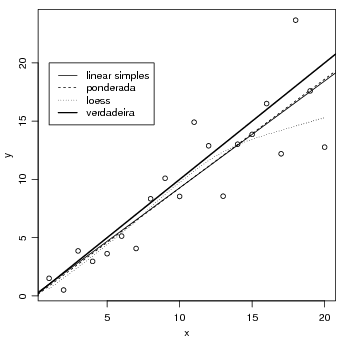
Fazendo uma regressão local não-paramétrica, e visualizando o resultado. Depois adicionamos a
linha de regressão verdadeira (intercepto 0 e inclinação 1), a linha da regressão sem ponderação e a
linha de regressão ponderada.
> lrf <- lowess(x, y)
> plot(x, y)
> lines(lrf, lty = 3)
> abline(coef(fm))
> abline(coef(fm1), lty = 2)
> abline(0, 1, lwd = 2)
> legend(1, 20, c("linear simples", "ponderada", "loess", "verdadeira"),
+ lty = c(1, 2, 3, 1), lwd = c(1, 1, 1, 2))
Ao final destas análises removemos o objeto dummy do caminho de procura.
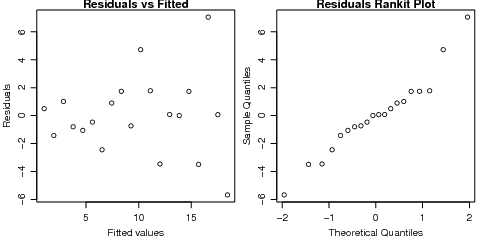
Agora vamos fazer um gráfico diagnóstico padrão para checar ajuste e pressupostos: o gráfico de
resíduos por valores preditos e gráfico de escores normais para checar assimetria, curtose e outliers
(não muito útil aqui).
> par(mfrow = c(1, 2))
> plot(fitted(fm), resid(fm), xlab = "Fitted values", ylab = "Residuals",
+ main = "Residuals vs Fitted")
> qqnorm(resid(fm), main = "Residuals Rankit Plot")
E ao final retornamos ao gráfico padrão e "limpamos"novamente o workspace, ou seja, apagando
objetos.
> par(mfrow = c(1, 1))
> rm(fm, fm1, lrf, dummy)
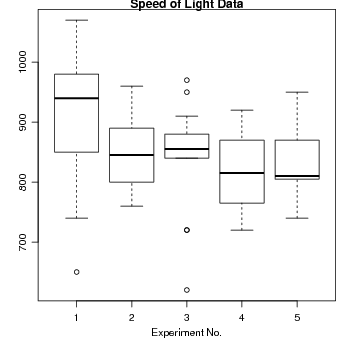
Agora vamos inspecionar dados do experimento clássico de Michaelson e Morley para medir a
velocidade da luz. Clique para ver o arquivo morley.tab de dados no formato texto. Se quiser voce
pode ainda fazer o download deste arquivo para o seu micro. Pode-se visualizar um arquivo externo
dentro do próprio R utilizando file.show() e note que no comando abaixo assume-se que o arquivo
está na área de trabalho do R, caso contrário deve ser precedido do caminho para o diretório
adequado.
> file.show("morley.tab")
Lendo dados como um "data-frame"e inspecionando seu conteúdo. Há 5 experimentos (coluna
Expt) e cada um com 20 “rodadas”(coluna Run) e sl é o valor medido da velocidade da luz numa
escala apropriada
> mm <- read.table("http://www.leg.ufpr.br/~paulojus/dados/morley.tab")
> mm
Expt Run Speed
001 1 1 850
002 1 2 740
003 1 3 900
004 1 4 1070
005 1 5 930
006 1 6 850
007 1 7 950
008 1 8 980
009 1 9 980
010 1 10 880
011 1 11 1000
012 1 12 980
013 1 13 930
014 1 14 650
015 1 15 760
016 1 16 810
017 1 17 1000
018 1 18 1000
019 1 19 960
020 1 20 960
021 2 1 960
022 2 2 940
023 2 3 960
024 2 4 940
025 2 5 880
026 2 6 800
027 2 7 850
028 2 8 880
029 2 9 900
030 2 10 840
031 2 11 830
032 2 12 790
033 2 13 810
034 2 14 880
035 2 15 880
036 2 16 830
037 2 17 800
038 2 18 790
039 2 19 760
040 2 20 800
041 3 1 880
042 3 2 880
043 3 3 880
044 3 4 860
045 3 5 720
046 3 6 720
047 3 7 620
048 3 8 860
049 3 9 970
050 3 10 950
051 3 11 880
052 3 12 910
053 3 13 850
054 3 14 870
055 3 15 840
056 3 16 840
057 3 17 850
058 3 18 840
059 3 19 840
060 3 20 840
061 4 1 890
062 4 2 810
063 4 3 810
064 4 4 820
065 4 5 800
066 4 6 770
067 4 7 760
068 4 8 740
069 4 9 750
070 4 10 760
071 4 11 910
072 4 12 920
073 4 13 890
074 4 14 860
075 4 15 880
076 4 16 720
077 4 17 840
078 4 18 850
079 4 19 850
080 4 20 780
081 5 1 890
082 5 2 840
083 5 3 780
084 5 4 810
085 5 5 760
086 5 6 810
087 5 7 790
088 5 8 810
089 5 9 820
090 5 10 850
091 5 11 870
092 5 12 870
093 5 13 810
094 5 14 740
095 5 15 810
096 5 16 940
097 5 17 950
098 5 18 800
099 5 19 810
100 5 20 870
Devemos definir Expt e Run como fatores tornar o data-frame visível na posição 2 do caminho de
procura.
> mm$Expt <- factor(mm$Expt)
> mm$Run <- factor(mm$Run)
> attach(mm)
Podemos fazer um gráfico para comparar visualmente os 5 experimentos
> plot(Expt, Speed, main = "Speed of Light Data", xlab = "Experiment No.")
Depois analisamos como um experimento em blocos ao acaso com Run e Expt como fatores e
inspecionamos os resultados.
> fm <- aov(Speed ~ Run + Expt, data = mm)
> summary(fm)
Df Sum Sq Mean Sq F value Pr(>F)
Run 19 113344 5965.5 1.1053 0.363209
Expt 4 94514 23628.5 4.3781 0.003071 **
Residuals 76 410166 5396.9
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
[1] "coefficients" "residuals" "effects" "rank" "fitted.values"
[6] "assign" "qr" "df.residual" "contrasts" "xlevels"
[11] "call" "terms" "model"
(Intercept) Run2 Run3 Run4 Run5 Run6
9.506000e+02 -5.200000e+01 -2.800000e+01 6.000000e+00 -7.600000e+01 -1.040000e+02
Run7 Run8 Run9 Run10 Run11 Run12
-1.000000e+02 -4.000000e+01 -1.000000e+01 -3.800000e+01 4.000000e+00 -1.754382e-13
Run13 Run14 Run15 Run16 Run17 Run18
-3.600000e+01 -9.400000e+01 -6.000000e+01 -6.600000e+01 -6.000000e+00 -3.800000e+01
Run19 Run20 Expt2 Expt3 Expt4 Expt5
-5.000000e+01 -4.400000e+01 -5.300000e+01 -6.400000e+01 -8.850000e+01 -7.750000e+01
Podemos redefinir o modelo, por exemplo ajustando um sub-modelo sem o fator “runs” e comparar
os dois modelos lineares via uma análise de variância.
> fm0 <- update(fm, . ~ . - Run)
> anova(fm0, fm)
Analysis of Variance Table
Model 1: Speed ~ Expt
Model 2: Speed ~ Run + Expt
Res.Df RSS Df Sum of Sq F Pr(>F)
1 95 523510
2 76 410166 19 113344 1.1053 0.3632
É importante saber interpretar os coeficientes segunda a parametrização utilizada. Por default a
parametrização é feita tomando o primeiro grupo como referência.
(Intercept) Expt2 Expt3 Expt4 Expt5
909.0 -53.0 -64.0 -88.5 -77.5
> mds <- tapply(Speed, Expt, mean)
> mds
1 2 3 4 5
909.0 856.0 845.0 820.5 831.5
2 3 4 5
-53.0 -64.0 -88.5 -77.5
E este comportamento é controlado por options(). Por exemplo, contrastes de Helmert são definidos
como se segue.
unordered ordered
"contr.treatment" "contr.poly"
> options(contrasts = c("contr.helmert", "contr.poly"))
> fm0 <- update(fm, . ~ . - Run)
> fm0$coef
(Intercept) Expt1 Expt2 Expt3 Expt4
852.400 -26.500 -12.500 -12.375 -5.225
> (2 * mds[3] - mds[1] - mds[2])/6
> (3 * mds[4] - mds[1] - mds[2] - mds[3])/12
> (4 * mds[5] - mds[1] - mds[2] - mds[3] - mds[4])/20
Enquanto que contrastes de cada tratamento contra a média geral são obtidos da forma:
> options(contrasts = c("contr.sum", "contr.poly"))
> fm0 <- update(fm, . ~ . - Run)
> fm0$coef
(Intercept) Expt1 Expt2 Expt3 Expt4
852.4 56.6 3.6 -7.4 -31.9
1 2 3 4 5
56.6 3.6 -7.4 -31.9 -20.9
Há algumas opções de contrastes implementadas no R e além disto o usuário pode implementar
contrastes de sua preferência. Para entender melhor os resultados acima analise as saídas dos
comandos abaixo.
2 3 4 5
1 0 0 0 0
2 1 0 0 0
3 0 1 0 0
4 0 0 1 0
5 0 0 0 1
[,1] [,2] [,3] [,4]
1 -1 -1 -1 -1
2 1 -1 -1 -1
3 0 2 -1 -1
4 0 0 3 -1
5 0 0 0 4
[,1] [,2] [,3] [,4]
1 1 0 0 0
2 0 1 0 0
3 0 0 1 0
4 0 0 0 1
5 -1 -1 -1 -1
.L .Q .C ^4
[1,] -6.324555e-01 0.5345225 -3.162278e-01 0.1195229
[2,] -3.162278e-01 -0.2672612 6.324555e-01 -0.4780914
[3,] -3.287978e-17 -0.5345225 2.164914e-16 0.7171372
[4,] 3.162278e-01 -0.2672612 -6.324555e-01 -0.4780914
[5,] 6.324555e-01 0.5345225 3.162278e-01 0.1195229
Se ainda não estivar claro experimente para cada uma destas examinar a matrix do modelo com os
comandos abaixo (saídas não são mostradas aqui).
> options(contrasts = c("contr.treatment", "contr.poly"))
> model.matrix(Speed ~ Expt)
> options(contrasts = c("contr.helmert", "contr.poly"))
> model.matrix(Speed ~ Expt)
> options(contrasts = c("contr.sum", "contr.poly"))
> model.matrix(Speed ~ Expt)
Ao final desanexamos o objeto e limpamos novamente o workspace.
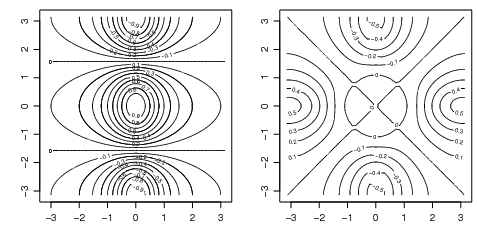
Vamos agora ver alguns gráficos gerados pelas funções contour() e image().
No próximo exemplo x é um vetor de 50 valores igualmente espaçados no intervalo [-pi pi]. y
idem. O objeto f é uma matrix quadrada com linhas e colunas indexadas por x e y respectivamente
com os valores da função cos(y)∕(1 + x2).
> x <- seq(-pi, pi, len = 50)
> y <- x
> f <- outer(x, y, function(x, y) cos(y)/(1 + x^2))
Agora gravamos parâmetros gráficos e definindo a região gráfica como quadrada e fazemos um mapa de
contorno de f. Depois adicionamos mais linhas para melhor vizualização. fa é a “parte
assimétrica” e t() é transposição. Ao final e restauramos os parâmetros gráficos iniciais.
> oldpar <- par(no.readonly = TRUE)
> par(pty = "s", mfrow = c(1, 2))
> contour(x, y, f)
> contour(x, y, f, nlevels = 15, add = TRUE)
> fa <- (f - t(f))/2
> contour(x, y, fa, nlevels = 15)
> par(oldpar)
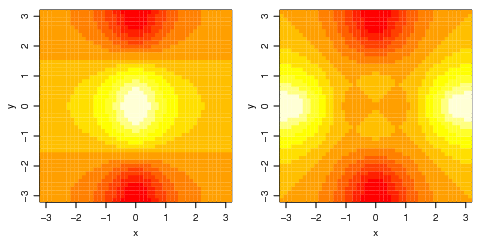
Fazendo um gráfico de imagem
> oldpar <- par(no.readonly = TRUE)
> par(pty = "s", mfrow = c(1, 2))
> image(x, y, f)
> image(x, y, fa)
> par(oldpar)
E apagando objetos novamente antes de prosseguir.
[1] "f" "fa" "mds" "mm" "oldpar" "x" "y"
Para encerrar esta sessão vejamos mais algumas funcionalidades do R. O R pode fazer operação com
complexos, note que 1i denota o número complexo i.
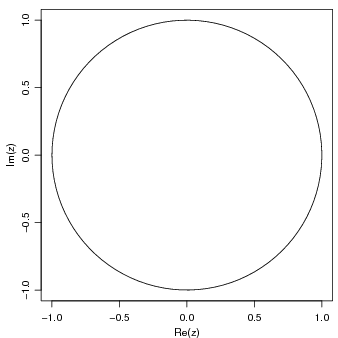
> th <- seq(-pi, pi, len = 100)
> z <- exp((0+1i) * th)
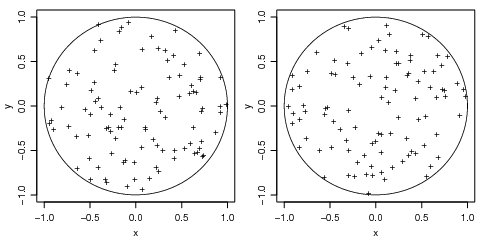
Plotando complexos significa parte imaginária versus real Isto deve ser um círculo: Suponha que
desejamos amostrar pontos dentro do círculo de raio unitário. uma forma simples de fazer isto é
tomar números complexos com parte real e imaginária padrão. E depois mapeamos qualquer externo
ao círculo no seu recíproco:
> par(pty = "s")
> plot(z, type = "l")
> w <- rnorm(100) + rnorm(100) * (0+1i)
> w <- ifelse(Mod(w) > 1, 1/w, w)
Desta forma todos os pontos estão dentro do círculo unitário, mas a distribuição não é uniforme.
Um segundo método usa a distribuição uniforme. os pontos devem estar melhor distribuídos sobre o
círculo
> plot(w, xlim = c(-1, 1), ylim = c(-1, 1), pch = "+", xlab = "x",
+ ylab = "y")
> lines(z)
> w <- sqrt(runif(100)) * exp(2 * pi * runif(100) * (0+1i))
> plot(w, xlim = c(-1, 1), ylim = c(-1, 1), pch = "+", xlab = "x",
+ ylab = "y")
> lines(z)
Apagamos novamente os objetos …
…e saímos do R.