Tabela de conteúdos
Histórico das aulas do curso
Ano 2014
Semana 1
- 14/08
- Manhã: apresentação, informações sobre o curso, percepções sobre geoestatística. Introdução ao programa R e ao pacote
geoR. Leitura de dados, formato geodata e análises exploratórias. - Tarde: dependência especial: correlogramas e variogramas. Interpolação espacial (krigagem) e mapas de predição. Outros formados de dados espaciais (dados de área e de processos pontuais).
- Atividades:
- Instalar o programa R e o pacote geoR
- Refazer o exemplo trabalhado em sala de aula
- Arquivo de dados (formato texto)
- Verificar outros conjuntos de dados disponíveis no pacote
geoRcom o comandodata(package="geoR")
e efetuar análises descritivas atentando as características a serem observadas comentadas em aula (distribuição e assimetria, dados atípicos, tendências com coordenadas e covariáveis, possível padrão espacial) - Obter outro conjunto de dados geoestatísticos (seu próprio ou em alguma fonte - web etc), carregar no R e efetuar análises
- Reproduzir e discutir as análises feitas com os comandos a seguir:
- 15/08
- Manhã: atividades computacionas
- Tarde: modelos com covariaveis - tendências com coordenadas e com covariáveis
- arquivos utilizados:
Semana 2
- 21/08
- Discussão sobre variogramas
- Extensões dos modelos considerando covariáveis e transformações
- Apresentação e discussão de trabalhos, dados e projetos de participantes
- 22/08
- Manhã
- Exemplo de código básico para predição espacial (arquivo de comandos), incluindo exportação dos resultados
-
- arquivo de dados utilizados no arquivo de comandos
- Tarde
- além de krigagem: Simulações da preditiva (condicionais)
Semana 3
- 18/09
- Manhã:
- simulação e funções de correlação
- estimação por verossimilhança
- Tarde
- Predição espacial e efeitos do modelo na predição
- inferência e predição Bayesiana
- 19/09
- Manhã:
- arquivo com comandos da aula do dia anterior - revisão, alterar e expandir os comandos
- Mais simulações: arquivo 2 simulações do modelo geoestatístico básico no R, feitas de várias formas
- arquivo de comandos de uma análise incluindo inferência Bayesiana
- Tarde:
- Modelos não gaussianos.
- arquivo 5 Simulação de modelos GLM geoestatísticos (Poisson e Binomial)
- Análises usando a geoRglm:
- arquivo com código para exemplo de simulação e análise de dados binários em 1-D
- arquivo de dados binários para análise com geoRglm
- Representação de dados espaciais no R pelo pacote sp. SpatialPolygons e algumas visualizações e análises de dados de áreas
- Convertendo um objeto de áreas/polígonos em geodata (centroides como coordenadas)
- Montar geodata a partir da importação de dados de centroides de municípios do Paraná
Semana 4
- 09/10: Estudos de caso, extensões e outras análises (processos pontuais e áreas)
- Manhã:
- SKATER
- Análise espacial de experimentos
- Dados composicionais (frações de solo)
- {http://www.ij-healthgeographics.com/content/10/1/17|Padrão espacial na resistência a antibióticos - EUREQA}
- Tarde:
- Abundância por classes de idade (pescada)
- Zona de captura
- Relação de variáveis de solo e ocorrência de espécies (Djair)
- Processos pontuais (princípios e conceitos básicos)
- 10/10
- Tarde:
- Apresentação e discussão de análises, temas e projetos de participantes do curso
- Modelo/código para Análise conjunta de diversas áreas
Ano 2012
Semana 1
- 13/09
- Manhã:
- Instalar o R e depois instalar o pacote adicional geoR (
install.packages("geoR", dep=T)) - Fazer o download para um diretório (criar um) de trabalho do arquivo de dados e do arquivo de comandos
- Rodar os comandos do arquivo, interpretar e discutir os resultados
- Discussão dos passos da análise: estrutura e importação dos dados, gráficos exploratórios e sua interpretação
- Pontos para atenção: dados atípicos (globais e locais), natureza da resposta e eventual necessidade de transformação, tendências no comportamento das variáveis.
- Tarde:
- Discussão dos passos da análise (cont).: variogramas (empíricos) e sua interpretação
- Ajuste de modelos a variogramas
- Interpolação espacial e visualização dos mapas
- 14/09
- Manhã:
- Estatística espacial: tipos "básicos" de problemas e estruturas de dados (dados de áreas, geoestatísticos e de processos pontuais). Semelhanças e diferenças. Estratégias para análise e modelagem.
- Exemplos de dados e problemas "geoestatísticos", estratégias de modelagem.
- Tarde:
- Exemplos de dados e problemas "geoestatísticos", estratégias de modelagem. (cont)
- Formalização e notação para modelagem geoestatística.
- Materiais
- Apresentação da primeira semana de aula sobre tipos básicos de problemas de estatística espacial
- Leitura de revisão: Cap 1 e 2 de Diggle & Ribeiro
- Arquivos de comandos com exemplos de análises (examinar antes da próxima aula)
-
- Comandos vistos em aula para porosidade total
- Outro arquivo de comandos para estes dados
- arquivo de dados de teores de elementos em camada 0-20acacio-artigo1.pdf
- arquivo com teores de elementos químicos em amostras de solo
-
- Sugestão de leitura Cap 3 de Diggle & Ribeiro
Semana 2
- 20/09
- Manhã: Caracterização dos modelos geoestatísticos e funções de correlação
- Tarde:
- Relações entre funções de correlação e trajetória dos processos,
- Variograma e função e correlação
- Simulação de processos
- Arquivos utilizados em anos anteriores (revisar/rodar)
- Um arquivo de comandos com exemplos introdutórios de simulação no R (modelos não espaciais)
- arquivo 2 simulações do modelo geoestatístico básico no R, feitas de várias formas
- arquivo de comandos 1 Um breve tutorial de uso da função
grf() - arquivo 5 Simulação de modelos GLM geoestatísticos (Poisson e Binomial)
- Definir e montar um conjunto de simulações. Montar o seu arquivo de comandos.
- Arquivos editados nas aulas
- Tarefas
- Refazer análises do arquivo usado na 1a semana da aulas em outro programa (outro pacote do R ou outro software)
- Alunos estatística (compulsório) / outros cursos (opcional)
- Escrever verossimilhança para estimar parâmetros com variâncias diferentes em diferentes áreas
- Escrever verossimilhança o modelo básico com uma função de correlação de suporte compacto (alcance finito) utilizando algoritmos de matrizes esparsas para operar com a matriz de covariâncias (ver pacote
sparseM) - Verossimilhança para distribuição t-multivariada
- Verossimilhança composta (Naimara)
Semana 3
- 04/10
- Manhã:
- Anisotropia e estacionaridade - extensões do modelo
- Predicões e simulações condicionais
- Predicao de funcionais
- arquivo de comandos básico do curso foi atualizado
- Tarde
- Discussão e temas variados
- Aula interrompida devido à falta de energia elétrica
- 05/10
- Manhã: Representação e dados espaciais
- Convertendo um objeto de áreas/polígonos em geodata (centroides como coordenadas)
- Montar geodata a partir da importação de dados de centroides de municípios do Paraná
- Tarde:
- Inferência e predição Bayesiana
- arquivo de comandos básico do curso foi atualizado
Semana 3
- 04/10
- Manhã:
- Revisão GLM. Simulações do GLM/GLGM
- arquivo de comandos de simulações
- Simulação de modelos GLM geoestatísticos(Poisson e Binomial)
- Análises usando a geoRglm:
- arquivo com código para exemplo de simulação e análise de dados binários em 1-D
- arquivo de dados binários para análise com geoRglm
Ano 2011
Semana 1
- 18/08
- Manhã:
- Apresentação e Introdução ao Sistema Estatístico R.
- Motivação para geoestatística: interpolação, alternativas, algoritmos, modelos determinísticos e estocásticos
- Tarde:
- Exemplo da aula: um script com os passos básicos de uma análise geoestatística canônica
- Discussão dos resultados do script. Passos da análise: (i) preparação dos dados, (ii) análise exploratória (distribuição, tendências, dados discrepantes globais e locais), (iii) modelos/inferência/estimação, (iv) predição (krigagem)
- 19/08
- Manhã:
- Lendo e analisando dados (experimentar com os seus dados e/ou com os dados a seguir)
-
- Comandos vistos em aula para pororidade total
- Outro arquivo de comandos para estes dados
- arquivo de dados de teores de elementos em camada 0-20
- arquivo com teores de elementos quimicos em amostras de solo
-
- Passos da análise de dados: preparação, exploratória, modelagem/estimação, predição
- Tarde:
- Lendo dados e análise geoestatística dados
- Tipos de dados espaciais: variação contínua (geoestatístico), discreta (areas) e processos pontuais
- Exemplo de alguns comandos para análise de dados espaciais de direrentes tipos
Atividades:
- Ler Capítulos 1 e 2 de Diggle e Ribeiro (2007)
- Selecionar um artigo que utilize métodos geoestatísticos e preparar uma resenha
- revisar, alterar e experimentar com os scripts vistos em aula
- Montar geodata a partir da importação de dados de centroides de municípios do Paraná
- Script de análise
- Convertendo um objeto de áreas/polígonos em geodata (centroides como coordenadas)
- tabela de atributos tabe.csv
Atividades:
- Examinar a página de tutoriais da geoR. Rodar os seguintes tutoriais:
Semana 2
- 25/08
- Manhã:
- Montando geodata passo a passo (arquivo de comandos)
- Problemas e alternativas de modelagem geoestatística
- Tarde:
- Análises de dados
- Modelo geoestatístico básico e extensões
- 16/08
- Manhã:
- Características e elementos do modelo geoestatístico e funções de correlação
- Tarde:
- exemplos
- Representação de objetos espaciais e fundamentos do pacote
sp
Semana 3
- 15/09
- Manhã:
- Modelos geoestatísticos, características, estacionariedade, anisotropia, simulações
- Variogramas teóricos e experimentais. Estimação por variograma dos parâmetros do modelo. Características e observações sobre o método de estimação
- Tarde:
- Praticando Simulação
- Um arquivo de comandos com exemplos introdutórios de simulação no R (modelos não espaciais)
- arquivo 2 simulações do modelo geoestatístico básico no R, feitas de várias formas
- arquivo de comandos 1 Um breve tutorial de uso da função
grf() - arquivo 5 Simulação de modelos GLM geoestatísticos (Poisson e Binomial)
- Definir e montar um conjunto de simulações. Montar o seu arquivo de comandos.
- Métodos de estimação: máxima verossimilhança
- 16/09
- Manhã:
- Modelos geoestatísticos, simulações em alguns modelos estatísticso e geoestatísticos
- Predição espacial e krigagem. Esperança, variância e simulações condicionais. Estimação de funcionais
- Tarde:
- Exemplo de código básico para predição espacial (arquivo de comandos), incluindo agora exportação dos resultados
- Inferência e predição Bayesiana para modelos geoestatísticos Gaussianos
- Ver e reproduzir os comandos abaixo da aula da Semana 3 de 2010 sobre predição, simulação condicional e análise Bayesiana
Semana 4
- 23/09
- Manhã: Revisão GLM. Simulações do GLM/GLGM
- arquivo de comandos de simulações
- Simulação de modelos GLM geoestatísticos(Poisson e Binomial)
- Tarde:
- arquivo com código para exemplo de simulação e análise de dados binários em 1-D
- arquivo de dados binários para análise com geoRglm
Semana 5
- 20/10
- Manhã e parte da tarde:
- tarde:
- fundamentos de análise de procesos pontuais
- apresentação alunos (ver material)
- 20/10
- Manhã:
- fundamentos de análise de dados de áreas
- algumas aplicações de geoestatística: análise espacial de experimentos e ánalises conjuntas
- tarde:
- interpolações não espaciais (arquivo de comandos com exemplos e alternativas)
- apresentação alunos (ver material)
Ano 2010
Semana 1
- 16/09
- Manhã:
- Tarde:
-
- 17/09
- Manhã:
- arquivo de dados de teores de elementos em camada 0-20
- arquivo de comandos editado na aula da tarde
- Tarde
- Análise de Fosforo mostrada na aula
- arquivo com teores de elementos quimicos em amostras de solo
Semana 2
- 23/09
- Scripts de simulação de Processos Gaussianos e de Dados geoestatísticos
- Simulação de dados não Gaussianos (modelo linear generalizado geoestatístico
- 24/09
- script modificado do dia 24/09 (atualizado, atualizar também a instalação da geoR)
- Tarde : Exemplos de Importação de Dados(Ricardo Olinda)
- Tarefa 1 : montar geodata a partir da importação de dados de centroides de municípios do Paraná
- Arquivos de dados:
Semana 3
- 14/10
- Simulações condicionais e predição de funcionais
require(geoR) ml <- likfit(s100, ini=c(1, 0.3)) gr <- expand.grid(seq(0,1,len=50), seq(0,1, l=50)) args(output.control) ## definindo simulacoes nos resultados (output) OC <- output.control(n.pred=1000, simulations.pred=T) kc <- krige.conv(s100, loc=gr, krige=krige.control(obj.m=ml), out=OC) ## vendo o que tem nos resultados names(kc) str(kc) ## os simulacoes ficam armazenadas aqui dim(kc$simulations) ## calculando (predizendo) FUNCIONAIS ## FUNCIONAL 1: mapa de probabilidade do atributo estar acima de 1,8 p1.8 <- apply(kc$simulations, 1, function(x) mean(x>1.8)) length(p1.8) image(kc, val=p1.8) ## adicionando legenda args(legend.krige) legend.krige(x.leg=c(1.05, 1.1), val=p1.8, y.leg=c(0.2, 0.8), vert=T) ## mudando os limites da image para incluir a legenda image(kc, val=p1.8, xlim=c(0, 1.2)) legend.krige(x.leg=c(1.05, 1.1), val=p1.8, y.leg=c(0.2, 0.8), vert=T) ## Outro funcional: proposção da área com valores acima de 1,8 A1.8 <- apply(kc$simulations, 2, function(x) mean(x>1.8)) length(A1.8) hist(A1.8, prob=T) lines(density(A1.8)) rug(A1.8) summary(A1.8) ## outro funcional : distribuição dos máximos sobre a área MAX <- apply(kc$simulations, 2, function(x) max(x)) length(MAX) hist(MAX, prob=T) lines(density(MAX)) rug(MAX) ## probabilidade do MAX está acima de 4 mean(MAX > 4) ## idem para minimo MIN <- apply(kc$simulations, 2, function(x) min(x)) summary(MIN) hist(MIN, prob=T) lines(density(MIN)) rug(MIN) ## ## um outro funcional diferente do anterior seria um mapa de maximos POR PIXEL MAX.map <- apply(kc$simulations, 1, function(x) max(x)) length(MAX.map) image(kc, val=MAX.map)
- 15/10
- Analise Bayesiana
## require(geoR) sata(s100) args(krige.bayes) args(model.control) MC <- model.control() args(prior.control() ## ## definindo prior para beta e fixando os valores dos demais parâmetros ## PC <- prior.control(beta="flat", sigmasq.pr="fixed", sigmasq=1, phi.prior="fixed", phi=0.3) ## definindo grid de predição gr <- as.matrix(expand.grid(seq(0,1,len=50), seq(0,1,len=50))) ## obtendo posterioris e preditivas s100.kb <- krige.bayes(s100, loc=gr, model=MC, prior=PC) ## inspecionando o output names(s100.kb) names(s100.kb$posterior) ## vendo os resultados da posterioris s100.kb$posterior ## e as predicoes na preditiva... names(s100.kb$predict) s100.kb$predict$mean[1:10] s100.kb$predict$var[1:10] s100.kb$predict$dist image(s100.kb, col=terrain.colors(21)) image(s100.kb, val=sqrt(s100.kb$predict$var), col=terrain.colors(21)) ## mapa de um funcional: probabilidade de estar acima de 2.0 ## calculando as probabilidades p2.0 <- pnorm(2.0, s100.kb$predictive$mean, sqrt(s100.kb$predictive$variance), low=F) ## e colocando no mapa image(s100.kb, val=p2.0, xlim=c(0, 1.2)) args(legend.krige) legend.krige(x.leg=c(1.05, 1.15), y.leg=c(0.2, 0.8), p2.0, vert=T, off=0.3) image(s100.kb, val=p2.0, xlim=c(0, 1.2), col=terrain.colors(5)) ## ## priori para beta e sigmasq ## args(prior.control) PCsig <- prior.control(beta="flat", sigmasq.pr="rec", phi.prior="fixed", phi=0.3) s100.kb.sig <- krige.bayes(s100, loc=gr, model=MC, prior=PCsig) names(s100.kb.sig) s100.kb.sig$posterior names(s100.kb.sig$predictive) ## pribalilidade na t (tem que corrigir o comando abaixo!!!!! p2.0 <- pt(2.0, s100.kb$predictive$mean, sqrt(s100.kb$predictive$variance), low=F) s100.kb.sig$predictive$dist ## ## prioris em beta, sigmasq e phi ## args(prior.control) PCphi <- prior.control(beta="flat", sigmasq.pr="rec", phi.prior="rec", phi.disc=seq(0, 1.5, by=0.1)) ## a chamada seria como abaixo (mas pode demorar muito para fazer predicao ## em um grid muito fino s100.kb.phi <- krige.bayes(s100, loc=gr, model=MC, prior=PCphi) ## definindo um grid mais "grosseiro" para teste gr <- as.matrix(expand.grid(seq(0,1,len=30), seq(0,1,len=30))) s100.kb.phi <- krige.bayes(s100, loc=gr, model=MC, prior=PCphi) names(s100.kb.names) names(s100.kb.phi) s100.kb.sig$posterior s100.kb.phi$posterior names(s100.kb.phi$posterior) s100.kb.phi$posterior$beta s100.kb.phi$posterior$sigmasq s100.kb.phi$posterior$phi names(s100.kb.phi$posterior) s100.kb.phi$posterior$sample ## visualizando as posterioris (marginais) ## beta|y hist(s100.kb.phi$posterior$sample[,1], prob=T) density(s100.kb.phi$posterior$sample[,1]) lines(density(s100.kb.phi$posterior$sample[,1])) rug(s100.kb.phi$posterior$sample[,1]) ## sigmasq|y hist(s100.kb.phi$posterior$sample[,2], prob=T, main=expression(sigma^2)) lines(density(s100.kb.phi$posterior$sample[,2])) rug(s100.kb.phi$posterior$sample[,2]) ## phi|y barplot(table(s100.kb.phi$posterior$sample[,3])) ## grafico "automatico" da geoiR com priori e posteriori plot(s100.kb.phi) ## experimentando com diferentes prioris ## note que aqui nao vamos fazer predição!!! PCphi <- prior.control(beta="flat", sigmasq.pr="rec", phi.prior="unif", phi.disc=seq(0, 1.5, by=0.05)) s100.kb.phi <- krige.bayes(s100, model=MC, prior=PCphi) plot(s100.kb.phi) ## PCphi <- prior.control(beta="flat", sigmasq.pr="rec", phi.prior="squar", phi.disc=seq(0, 1.5, by=0.05)) s100.kb.phi <- krige.bayes(s100, model=MC, prior=PCphi) plot(s100.kb.phi) ## priori com amis pontos na discretizacao PCphi <- prior.control(beta="flat", sigmasq.pr="rec", phi.prior="unif", phi.disc=seq(0, 1.5, by=0.05)) s100.kb.phi <- krige.bayes(s100, model=MC, prior=PCphi) plot(s100.kb.phi) ## especificando uma priori particular do usuário args(dgamma) curve(dgamma(x, 2, sc=0.05), from=0, to=1.5) curve(dgamma(x, 2, sc=0.1), from=0, to=1.5) curve(dgamma(x, 2, sc=0.15), from=0, to=1.5) ## discretizando PRIORphi <- dgamma(seq(0, 1.5, by=0.05), 2, sc=0.15) ## .. e garantindo que soma 1 na discreta PRIORphi <- PRIORphi/sum(PRIORphi) PCphi <- prior.control(beta="flat", sigmasq.pr="rec", phi.prior=PRIORphi, phi.disc=seq(0, 1.5, by=0.05)) s100.kb.phi <- krige.bayes(s100, model=MC, prior=PCphi) plot(s100.kb.phi)
Semana 4
- 28/10
- Manhã:
- Tarde:
- Análise do GLGM (dados binários e Poisson)
- 29/10
- Modelo Bivariado script da aula
- Tutoriais geoR:
Ano 2009
Semana 1
- 17/09
- Manhã:
- Tarde:
- arquivo de dados de teores de elementos em camada 0-20
- arquivo com teores de elementos quimicos em amostras de solo
- arquivo de comandos editado na aula da tarde
- 18/08
- Manhã:
- Efetuar análises com variáveis dos dados dos arquivos acima. Escolher alguns arquivos e variáveis procurando fazer análises adequadas.
- Depois de ter tentado efetuar as análises mencionadas acima, experimente reproduzir os comandos dos arquivos a seguir. Reflita sobre os comandos utilizados e discuta os resultados.
- Arquivo de comandos (análise do fósforo (P) de
profund020a.txt - Arquivo com comandos do R com análise geoestatística utilizados na aula
Semana 2
- 24/09
- Manhã: Propriedades dos modelos geoestatísticos e funções de correlação
- Tarde: Propriedades dos modelos e simulação de campos aleatórios Gaussianos
- 25/09
- Manhã: Estimação de parâmetros: variogramas e verossimilhança
- Tarde: Predição espacial (krigagem)
Semana 3
- 22/10
- Manhã: prática e Explorando a predição espacial: simulação condicional e predição de funcionais genéricos.
- arquivo de comandos: análise de ctc40
- Dados:
- Tarde: prática e Análise espacial de experimentos
- 23/10
- Manhã: Inferência Bayesiana para modelos geoestatísticos
- Tarde: Integração com ambientes de SIG e p pacote aRT
Semana 4
- 29/10
- Manhã: Revisão de análise Bayesiana do modelo geoestatístico
- Tarde: Modelos Multivariados e Modelos para dados composicionais
- simulação de dados bivariados
- estimação e predição de dados bivariados
- arquivo com comandos para análise dos dados composicionais
- 30/10: Modelo Linear Generalizado Geoestatístico
- arquivo com código para exemplo de simulação e análise de dados binários em 1-D
- arquivo com código para exemplo de simulação e análise de dados de contagem (Poisson)
Ano 2008
Semana 1
- Aula 1 (01/09/2008):
- Manhã: introdução a estatística espacial. Análise espacial, estatística espacial e geoestatística
- Transparências do curso, Session 1 Apresentação da primeira semana
- página com comandos ilustrando algumas funcionalidades de estatística espacial
- tarde: tipos de problemas e estruturas de dados em geoestatística
- Transparências do curso, Session 2
- Aula 2 (02/09/2008): introdução à análise geoestatística de dados
- Arquivo de dados usado na aula (manhã)
- Arquivo de comandos (análise do fósforo (P) de
profund020a.txt - Arquivo com comandos do R com análise geoestatística utilizados na aula (2007)
Semana 2
- Aula 3 (08/09/2008)
- Uma visão de geoestatística baseada em modelos
tranparências do curso, Session 3
- Aula 4 (09/09/2008)
- Manhã: propriedades do modelo geostatístico e extensões. Simulação de dados
transparâncias do curso Session 4 & 5 - Tarde:
- extensões do modelo básico
transparâncias do curso Session 5 (cont.) - Estimação: variogramas e verossimilhança. Cross-validação. Escolha de modelos e critérios. Estudos de caso.
transparências do curso Session 6.
- Atividade: para próxima aula deve-se:
- entregar o artigo e a resenha sobre ele. pode ser impresso ou eletrônico
- é altamente desejável que já tenham o banco de dados com análises feitas. Não compulsória a entrega ainda mas será para próxima aula. Na próxima aula devem discutir qq dúvida referente aos dados comigo e colegas.
Semana 3
- Aula 5 (29/09/2008): predição espacial - krigagem. Propriedades, algorítmos e exemplos
- Transparências do curso, Session 7: Fundamentos da interpolação espacial (krigagem)
- arquivo de comandos utilizado na aula
- Aula 6 (30/09/2008)
- Dados: regioes.dat Estendendo a predição espacial
simulações condicionais e predição de funcionais genéricos. Predição Bayesiana e Análise espacial de experimentos - arquivo de comandos: análise de ctc40
Semana 4
- Aula 7 (20/10/2008): GLGM
- Transparências do curso, Session 9: GLGM
- arquivo de comandos para simulação de GLGM
- Aula 8 (20/10/2008): GLGM
- Transparências do curso, Session 10: GLGM - estudos de caso
- Simular dados nas coordenadas as estações meteorológicas do Paraná (geoR:
data(parana)) segundo o GLGM Poisson de média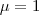 ,
, 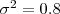 ,
, 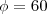 e offset pelos dados no arquivo (mesma ordem das coordenadas das estações):
e offset pelos dados no arquivo (mesma ordem das coordenadas das estações):- arquivo com valores de offset para simulação
- Analisar os o conjunto de dados simulados usando a geoRglm obtendo inferências sobre os parâmetros e predição da intensidade no estado
Semana 5
- Aula 9 (03/11/2008): GLGM e extensões do modelo geoestatístico
- Transparências do curso, Session 9: GLGM - comentários adicionais sobre implementação do algorítimo MCMC na geoRglm
- Estudos de caso, RAPLOA: Transparências do curso, Sessão 10
- Extensões do GLGM: variogramas e processo pontual Log-Cox Gaussiano, Transparências do curso, Sessão 11
- Processos pontuais marcados e amostragem preferencial, Transparências do curso, Sessão 11
- artigo de Schlather, Ribeiro e Diggle sobre teste de independência entre marcas e pontos
- artigo de Yongtao Guan sobre o mesmo tema
- Delineamentos geoestatísticos, Transparências do curso, Sessão 11
- Simular processos pontuais:
(a)Poisson homogêneo,(b)Poisson não homogêneo,©Log-Cox Gaussiano. No ultimo caso verificar o efeito dos parâmetros do modelo. - Simular processos pontuais marcados:
(a)com marcas e pontos independentes,(b)marcas e pontos dependentes - Aula 10 (04/11/2008): Extensões do modelo geoestatístico
- modelos espaço temporais - opções e abordagem utilizando funções de covariância espaço temporais. Um exemplo de aplicação
- combinando geoestatística e análise de dados composicionais – um exemplo na avaliação de estoque de peixes
- Modelos geoestatísticos multivariados. Construção de modelos, opções e o BGCCM
- Classes para dados espaciais no pacote sp
Outros conjuntos de dados e arquivos de comandos
- dados de características de solo (arquivo de dados)
- precipitação no PR em 1993 (arquivo de dados)
- propriedades físicas do solo (arquivo de dados)
- porosidade e outras características do solo a 30 cm (arquivo de dados)
- Arquivo com comandos do R com análise geoestatística utilizados durante aula
- outro arquivo com comandos do R com análise geoestatística utilizados durante aula